読んだ。
Optimizing Prone Cardiopulmonary Resuscitation: Identifying the Vertebral Level Correlating With the Largest Left Ventricle Cross-Sectional Area via Computed Tomography Scan.
Anesth Analg. 2017 Feb;124(2):520-523.
CTでの左心室最大断面積は、肩甲骨下縁の0-2椎体下で86% くらいの人が該当するから、ここを押しておけばどうにかなる。
既報では伏臥位でのCPR は、仰臥位でのCPR と同等もしくはそれ以上の駆出効率があるとかなんとからしく、仰臥位に戻せない脊椎手術、開頭術などではここをメルクマークにがんばる。ただし、手術室で気道確保がされていることが前提なので、市中で遭遇した場合は普通にひっくり返したほうがいいと思う。
googlevis でいろいろ触れるプロットを作る

この記事はR Advent Calendar 2018 の24日目の記事です。
R でプロットするときに、標準ではgraphics のなかにあるplot を使ってべたなプロットを作ると思うが、最近ではggplot なんかが流行っている。
最近、プロット上にマウスカーソルを持ってくるとデータの内容を確認できる、html ベースの我らがgoogle 様のおつくりになったgooglevis というパッケージが使えるような、いややっぱ使えないような感じだったので紹介する。
とりあえずやってみると、自分のデフォルトで設定しているブラウザーでナイル川の流量サンプル折れ線グラフができる。
library(googleVis) dat <- data.frame(year=seq(tsp(Nile)[1], tsp(Nile)[2]), flow=Nile@.Data) opt <- list(lineWidth=3, width=800, height=300, legend="none", title="Nile flow", vAxis="{title: 'Flow'}", hAxis="{title:'Year'}") g <- gvisLineChart(dat, xvar="year", yvar="flow", options=opt) plot(g)
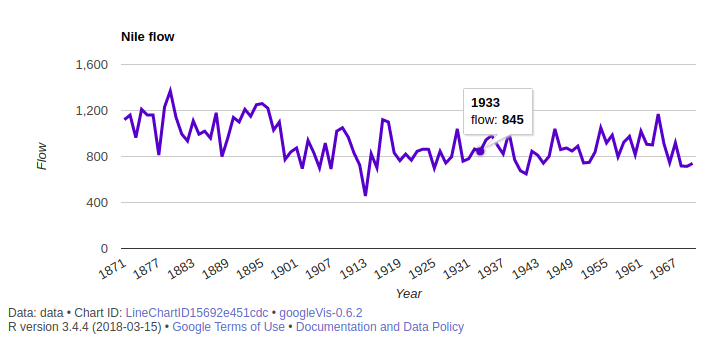
ここで、作成したオブジェクト g は、html である。
R で描出する場合は、
plot(g)
で表示されるが、このhtml を流用すると、R 以外で使えるようになる。
ちなみに、プロットの下にある
Data: data • Chart ID: LineChartID15692e451cdc • googleVis-0.6.2 R version 3.4.4 (2018-03-15) • Google Terms of Use • Documentation and Data Policy
が邪魔な場合は、g の中のhtml を削除すればよい。具体的には
g$html$caption <- NULL g$html$footer <- NULL
で消える。
例えば、このhtml をそのままサイトに貼り付けると、googlevis のAPI が勝手に理解して、サイト上でグラフにしてくれる。
はてなブログであれば、記事を書くときに「HTML 編集」モードがあるので、そこにベタ貼りして、プレビューすると、html 表示でgooglevis のグラフを見ることができる。ただし、HTML 編集モードで書き始めてしまった記事は、その後HTML 編集モードでないと編集できない仕様らしいので、HTML タグが分からない場合にお手軽記法が使えないと思うので少しハードルが高い。
おそらくQiita やgithub でもできそうな気がする。
はてなダイアリーではちょっと頑張ってもできなかったのであきらめた。どのみち2019年春に使えなくなるらしいのではてなブログへの移行をする時期なのか...
2019年春「はてなダイアリー」終了のお知らせと「はてなブログ」への移行のお願い - はてなダイアリー日記
googlevis が使いにくい点は、グラフをいじりたいときにgooglevis が定める仕様以外のことはなかなかしにくいことだと思う。
こちらを参考にパラメータをいじればどうにかなる。
サイトにHTML ベースの触れるプロットを埋め込む方法は他にもあって、例えば水平ラベルをいじりたいときに、hAxis をいじるわけだが、hAxis にはhAxis.color やhAxis.gridlines があり、さらにhAxis.gridlines にはhAxis.gridlines.color やhAxis.gridlines.count などが定められている。これらをリスト形式でひたすらoptions=... にいれるわけだが、例えば色を複数設定したいときには色の数と、どの範囲の値のときにその色を対応させるか、などを記述しないといけない。これがひたすらhoge.color に記載されないと色を読み込んでくれないわけだが、手打ちするのは面倒なのでそこはsprintf 関数などを使って機械的にテキスト処理するとなんとかなる。けっこう面倒だった。
あとネットにつながっていないとプロットされない。

html ベースの、いろいろ触れるプロットを作るには他にも方法があって、例えばplotly はその候補である。これはオフラインでも描出できるし、ブログにも貼り付け可能だが、ユーザー登録が必要(?) らしいので、何も考えずにhtml として貼り付けるならばgooglevis のほうがよさそうだった。
第113回医師国家試験を項目反応理論で解析します(予告)、の予備実験

予備実験用の解答サンプルシートはこちら
第112回から適当に20問とってきたもの。
↓
https://docs.google.com/forms/d/e/1FAIpQLSfiLVA-1m7NC5SMmyi0X1ftSYJzhWuOac6kr-WMR8gKgFLWVA/viewform
shiny 上でその20問の項目反応理論(IRT)
↓
https://yfujii08.shinyapps.io/medicalexamltm/
※重要な話なので先頭にもってきました※
データを集めるわけだが、某予備校や解答収集サービスのようにセキリュティ強固なサーバーを持っているわけではなくむしろ予算 ゼ ロ 円解析なので、グーグルアンケートフォームにひたすらみなさんの善意でポチポチやってもらえれば、と思っています。
予備実験では20問、たぶん1問1分くらいかかって15−20分くらい時間を使わせてしまいます。
本番では400問あるので、受験者が1万人、上記サービスで7000人くらい入力しているらしいので、ネット宣伝と口コミで希望者・入力者を募りつつ、サーバー負荷やセキリュティ問題はgoogle 様やshiny 様に丸投げしようと思います。個人の特定はできないようにします。
※以下から本文※
医師国家試験とは、2日間(昔は3日間)にわたって400問をマークシートで解く。
そのほとんどは5択から1つ正解を選べ、だが、2つ選べや3つ選べ、計算結果を数値を塗りつぶして答えろ、1問1点の場合と3点の場合がある、A-F の6ブロックある、一般/臨床問題と必修問題の両方を合格基準を超えないといけない、禁忌肢という選んでしまったらそれだけで不合格となる問題や選択肢がある、といったレギュレーションがある。
そこで受験生は、ひたすら問題を解くわけだが、合格率90% を誇る試験で、たいていの問題は正答率100% に近かったりするわけだが、割問という「2択ないしは複数選択肢に解答が割れる」という問題がある。
これが毎年話題になる。
受験生としては、自分が答えた問題の正答が気になるので、○ECとかTECO○とか○んコレとかに解答を登録したりするわけだが、ここらでいう割問というのは、「その問題単体で見た時の、解答のばらつき」しか見ていない。
さて、項目反応理論(IRT) というのは、マルチョイ問題において、受験者個人の能力値(すべての問題への正答率を考慮している)、問題の難易度(その個々人が、どの問題を正答できて、どの問題を誤答したかという情報を考慮する)といったパラメータを定量的に推定する。
ここで、mirt パッケージにあるSAT12 という試験解答サンプルを使ってみる。SAT12 は600人32問題の解答パターンをもつ。
これを正解したかしてないかの1/0 バイナリデータにする。IRT 自体はltm パッケージを使うことにして、IRT により各問題の難しさや識別能の推定はltm 関数を、各個人の能力はfactor.scores 関数で得られる。
# 先頭の15人、8問目まで Item.1 Item.2 Item.3 Item.4 Item.5 Item.6 Item.7 Item.8 1 1 4 5 2 3 1 2 1 2 3 4 2 8 3 3 2 8 3 1 4 5 4 3 2 2 3 4 2 4 4 2 3 3 2 4 5 2 4 5 2 3 2 2 1 6 1 4 3 1 3 2 2 3 7 1 4 5 2 2 2 2 2 8 2 4 1 5 3 2 2 5 9 5 1 2 3 2 2 4 4 10 5 3 4 2 3 2 4 5 11 2 1 4 5 2 3 2 4 12 3 1 3 4 2 2 2 2 13 4 5 5 4 2 2 4 1 14 1 4 3 2 3 2 2 5 15 3 4 3 5 5 2 2 2
library(ltm) library(mirt) key <- c(1,4,5,2,3,1,2,1,3,1,2,4,2,1,5,3,4,4,1,4,3,3,4,1,3,5,1,3,1,5,4,5) # 正解 data <- key2binary(SAT12, key=key) # バイナリ化 l <- ltm(data ~ z1) # IRT ans <- t(mapply(function(z) table(factor(SAT12[,z], 1:5)), 1:ncol(SAT12))) plot(l)
32問分の難しさはこんな感じで出力される。

さて、各問題について見てみる。32問分の解答分布、正答、IRTの推定値は以下のとおりである。
cbind(round(ans/nrow(SAT12), 3), key, l$coefficients)
1 2 3 4 5 key (Intercept) z1 Item.1 0.283 0.203 0.267 0.232 0.013 1 -1.0458445 0.8018208 Item.2 0.212 0.022 0.070 0.568 0.127 4 0.4361887 1.5040666 Item.3 0.165 0.183 0.260 0.098 0.280 5 -1.1425458 1.0746489 Item.4 0.165 0.378 0.148 0.172 0.128 2 -0.5309422 0.5845126 Item.5 0.093 0.143 0.620 0.093 0.048 3 0.6044634 0.9900492 Item.6 0.160 0.582 0.107 0.043 0.108 1 -2.0509622 1.1494988 Item.7 0.025 0.760 0.007 0.190 0.017 2 1.3818932 1.0042941 Item.8 0.202 0.205 0.207 0.250 0.133 1 -1.5090070 0.6927192 Item.9 0.065 0.010 0.885 0.033 0.007 3 2.1427139 0.5328306 Item.10 0.422 0.215 0.165 0.028 0.167 1 -0.3615247 1.0086219 Item.11 0.003 0.983 0.008 0.003 0.002 2 5.2441772 1.7296232 Item.12 0.072 0.082 0.218 0.415 0.205 4 -0.3455723 0.1611497 Item.13 0.110 0.662 0.070 0.118 0.040 2 0.8507691 1.1074704 Item.14 0.723 0.027 0.108 0.022 0.117 1 1.1727517 1.0370366 Item.15 0.035 0.062 0.060 0.025 0.817 5 1.9224593 1.2930864 Item.16 0.070 0.105 0.413 0.215 0.195 3 -0.3825038 0.7263422 Item.17 0.008 0.005 0.010 0.963 0.013 4 4.1603459 1.5479738 Item.18 0.303 0.033 0.165 0.352 0.142 4 -0.8530572 1.7008815 Item.19 0.548 0.053 0.358 0.030 0.010 1 0.2363424 0.8403153 Item.20 0.012 0.002 0.105 0.873 0.007 4 2.6034887 1.5311169 Item.21 0.050 0.008 0.915 0.013 0.012 3 2.5167030 0.6058270 Item.22 0.028 0.005 0.935 0.017 0.015 3 3.4736774 1.5369172 Item.23 0.290 0.177 0.128 0.313 0.087 4 -0.8505416 0.6379289 Item.24 0.728 0.162 0.042 0.022 0.045 1 1.2673661 1.2051755 Item.25 0.240 0.170 0.375 0.065 0.142 3 -0.5674927 0.7713010 Item.26 0.020 0.227 0.030 0.262 0.460 5 -0.1728073 1.5352022 Item.27 0.862 0.093 0.012 0.020 0.010 1 2.7575590 1.9045292 Item.28 0.082 0.010 0.530 0.337 0.037 3 0.1722115 1.0711318 Item.29 0.340 0.295 0.205 0.085 0.067 1 -0.7508683 0.8350190 Item.30 0.150 0.110 0.107 0.183 0.440 5 -0.2484688 0.3855356 Item.31 0.075 0.020 0.012 0.833 0.058 4 2.7738863 2.3285203 Item.32 0.125 0.183 0.443 0.075 0.162 5 -1.6516711 0.1291500
IRTの推定値をプロットするとこうなる。
i1 <- c(10, 12, 16, 26, 30) i2 <- c(2, 17, 20, 22, 26) plot(l$coefficient,type="n") abline(v=mean(l$coefficients[i1, 1]), lty=3, col="blue", lwd=3) abline(h=mean(l$coefficients[i2, 2]), lty=3, col="green", lwd=3) text(l$coefficients[,1], l$coefficients[,2], 1:ncol(SAT12), font=2)
似たようなintercept を持つがz1 が異なる10, 12, 16, 26, 30 番の問題を取り上げてみると(青の垂直線、下図のひだり側)、いずれも割問である。しかし、赤の12番が平たい曲線なのに比べ、青の26番はいくぶん急峻な曲線である。
これは、前者が高得点者でもそんなに得点率が上昇しないのに比べて、後者はある得点水準から得点率が急上昇するので、その境界線の能力の受験者を分けるのにはよい。
12番と26番を見てみると、どちらも正答率は40%半ばであるが、問題の性能としてはずいぶん違う。26番は良問だが、12番はクソ問。
1 2 3 4 5 key (Intercept) z1 Item.10 0.422 0.215 0.165 0.028 0.167 1 -0.3615247 1.0086219 Item.12 0.072 0.082 0.218 0.415 0.205 4 -0.3455723 0.1611497 Item.16 0.070 0.105 0.413 0.215 0.195 3 -0.3825038 0.7263422 Item.26 0.020 0.227 0.030 0.262 0.460 5 -0.1728073 1.5352022 Item.30 0.150 0.110 0.107 0.183 0.440 5 -0.2484688 0.3855356
似たようなz1 を持つがintercept が異なる2, 17, 20, 22, 26 番の問題を取り上げてみると(緑の水平線、下図のみぎ側)、どれも似たような急斜面の曲線だが、左右に位置がずれている。正答率を見ると、赤の17番の問題で96%、水色の26番の問題で46%であり、みぎにいくほど正答率が低い難問である。
1 2 3 4 5 key (Intercept) z1 Item.2 0.212 0.022 0.070 0.568 0.127 4 0.4361887 1.504067 Item.17 0.008 0.005 0.010 0.963 0.013 4 4.1603459 1.547974 Item.20 0.012 0.002 0.105 0.873 0.007 4 2.6034887 1.531117 Item.22 0.028 0.005 0.935 0.017 0.015 3 3.4736774 1.536917 Item.26 0.020 0.227 0.030 0.262 0.460 5 -0.1728073 1.535202

各問題の難易度や良問具合がわかったので、次に、受験者個人の成績や能力値をどう定量化するかであるが、これはfactor.scores という関数で得られる。ここから結果を見ると、32問題の正答/誤答パターンが600人中どれくらいあったか(Obs) というデータから、z1(正規化した分布における確率点)が推定されるので、ここが自分(の解答から得られる正誤パターンにおける)の能力値(同一試験を受けた他の受験者全員をひっくるめた集団での)になる。
SAT12 データセットでは、全問正解が3人いるようである。
ここからz1 を拾ってきて横軸を定め、元のIRT 曲線に垂線を引くと、自分の能力で各問題をどれくらいの確率で正答できたのかが確率的にわかる。
f <- factor.scores(l) f$score.dat
Item.1 Item.2 Item.3 Item.4 Obs Exp z1 se.z1 1 0 0 0 0 1 7.320352e-07 -1.125597 0.3501032 2 0 0 0 0 1 1.192118e-05 -1.035763 0.3510456 3 0 0 0 0 1 7.614205e-06 -1.268032 0.3503369 4 0 0 0 0 1 2.158900e-04 -1.578284 0.3585213 5 0 0 0 0 1 1.354704e-03 -2.044771 0.3858258 6 0 0 0 0 1 7.785685e-06 -1.299507 0.3506911 7 0 0 0 0 1 2.653074e-04 -1.303414 0.3507428 8 0 0 0 0 1 9.714099e-05 -1.137476 0.3500392 9 0 0 0 0 1 2.220848e-05 -1.500034 0.3555113 10 0 0 0 0 1 2.611964e-04 -1.573319 0.3583131 . . . 598 1 1 1 1 3 4.027146e-02 2.590108 0.6140229 ← 全問正解が Obs = 3
データはどう集めるか問題があるが、実は某サービスが始まったときにデータを打診したらゴニョゴニョがあったので今回の解析を断念していたが、google 様とshiny 様のお力を借りて出来そう、と思い立ったのでネットで宣伝してN数稼げたらいいなあ…と思う。冒頭に書いた。
昔は3日間あった試験が今では2日間しかないのだから勉強量は2/3 でいいでしょう?

昔、とある試験を受けたのだが、最近では改定されて3日間あった日程が2日間に減っているらしい。
ならば昔の受験生がいままで一生懸命勉強に費やしていた期間は今の人達は2/3 でいいんじゃね?(適当 と後輩を煽っていたのだが、以前試験勉強に飽きたので今までやった模擬試験の得点推移から本番の得点を予想しようと遊んだ話をしたのに、肝心の得点推移と解析結果を記事にしていなかったようである。
というわけで図だけとってきた。

17回分の模擬試験を受けてそのときどきの95% 信頼区間と、本番での得点予想は一般 0.750, 臨床 0.801 ,必修 0.879 だったようである。
自然科学研究のためのR入門: 再現可能なレポート執筆実践 (Wanderful R)
読んだ。

自然科学研究のためのR入門―再現可能なレポート執筆実践― (Wonderful R 4)
- 作者: 江口哲史,石田基広,市川太祐,高橋康介,高柳慎一,福島真太朗,松浦健太郎
- 出版社/メーカー: 共立出版
- 発売日: 2018/10/11
- メディア: 単行本
- この商品を含むブログ (1件) を見る
カラーになって3000円とはかなり破格の値段設定だと思う。
Rmd ファイルの内容と、実際のコンパイル結果が載っているのは、わかる/できる人からすれば冗長で紙面の無駄だが、正直なところ全部載せしないとわからない人というのは手を動かし始めのときなど特にそうなので、入門書としては良いと思う。
が、だからといって完全に初心者でもわかりやすいかというとそうでもないと思う。それなりにやってきてないと最初に読んだ時にはわからないと思う。Rとしてもコンピューターとしても生化学としても。
急速導入のセリック手技はしなくてもいいのではないか
読んだ。
Effect of Cricoid Pressure Compared With a Sham Procedure in the Rapid Sequence Induction of Anesthesia: The IRIS Randomized Clinical Trial.
JAMA Surg. 2018 Oct 17. doi: 10.1001/jamasurg.2018.3577.
緊急手術時の急速導入で、セリック手技するかしない(Shamという)で、挿管時の嘔吐リスクがどうなのかを非劣性試験で試したもの。
結論から言うとSham の非劣性は示せなかったので、この記事のタイトルだけを読んで「セリック手技しません」というのはやめておいたほうがいい。
ASAPS2 程度までの、なんのリスクのない虫垂炎や胆嚢炎は任されるようになったが、それでも導入時はフルストマックなので嘔吐、誤嚥のリスクはつきまとう。
手術場で嘔吐されたことはないが、研修医のときにERで若い男性のイレウスに胃管をいれたらドボドボ吐かれてクソ日和ったことと、バイト先で朝起きてこない高齢女性が息も絶え絶えでSpO2 80台で搬送されてきて、挿管しようと口を見たら真っ青で、挿管時にこれまたドボドボと青い液体を吐かれて、吸引してもズルズル青い液体が引けて犯人はロヒプノールでした、ということはあった。
というわけで、この論文では挿管時の嘔吐発生事前確率を2.8% に設定しているが、根拠とした論文は手術場以外のすべての挿管状況を含んでいるので、けっこう高い印象である。セリック手技は誤嚥を防ぐためにしたほうがいいと45年前に言われたらしいが、それを支持する強固な証拠というのがあるのかというとそうでもないらしい。成書には「してもしなくても…」とは書いてあったのをみたことはある。でも書かれている以上、やっておくのは無難だと思われる。
試験は有意水準0.05、検出力0.8で、両群それぞれ1700例程度が必要となった。primary outcome は挿管直後の誤嚥で、セリック手技では10/1784 (0.6%), Sham では9/1736 (0.5%) 例の誤嚥が生じて、Sham でも全然いけるやん、と思ったらRR は最大で2 になるため、非劣性は示せなかった。というのも、本文でリスクが95%CI 上限で1.5 を超えないときに、非劣性を示す、と定義したので、非劣性かどうかでいうと、統計学的には非劣性を示せなかったようである。これは事前確率を2.8% と設定したものの、実際に手術室で用意万端(プロポフォールと、ほとんどがサクシニルコリンで導入)の状態で挿管するので、誤嚥の発生は高く見積もり過ぎてた、ということらしい。
誤嚥するときの事前確率が0.6% だった場合に、非劣性を示すのにどれくらいの症例数が必要だったかというと、もともとの2.8% のときは1.5 倍(4.2%)までを許容する、と考えればこんな感じになる。ずれてはいるが2100人くらいは各群に必要、つまり4200人はリクルートされないといけない。
power.prop.test(p1=0.028, p2=0.028*1.5, sig.level=0.05, power=0.8, alternative="one.sided")
Two-sample comparison of proportions power calculation
n = 2129.728
p1 = 0.028
p2 = 0.042
sig.level = 0.05
power = 0.8
alternative = one.sided
NOTE: n is number in *each* group
0.6% の事前確率のときは、1.5倍して0.9% までを許容するので、差が小さいときには非常に大きなサンプルサイズを必要とする。各群で10000人は必要となる。
power.prop.test(p1=0.006, p2=0.006*1.5, sig.level=0.05, power=0.8, alternative="one.sided")
Two-sample comparison of proportions power calculation
n = 10225.93
p1 = 0.006
p2 = 0.009
sig.level = 0.05
power = 0.8
alternative = one.sided
NOTE: n is number in *each* group
Sham は非劣性だったけれども、secondary outcome としては、セリック手技したほうが挿管までに時間がかかる(27秒 vs 23秒)とか、結局セリック手技を中断するはめになって、セリック手技をやめたらCormack が改善した(62% vs 33%)とか、挿管すること自体に影響を与えるようである。初回挿管成功率はどちらの群も92% だった。
フランスの報告だが麻酔看護師が60% 程度挿管に携わっているようである。ほへぇ。
まともにセリック手技というものを教えてもらったことがないのであれだが、30N の圧力でthe first 3 fingers(親指、人差し指、中指のことでいいの?)を使ってC5 に向けて押す、ということらしい。論文では50ml シリンジを使って、40ml の空気を33ml にまで圧縮するくらいの押し具合がそうなるようで、これを使って教育したらしい。
ようやくShinyを使ってインタラクティブにパラメータをいじって遊べるシミュレーターをweb上にあげることができた

Shinyというhtml 上でいろいろパラメータをいじったものを即座に計算して描出するインタラクティブでなんかすごいアプリがあったのだが、rmarkdown と同様に手をつけることなく放置していた。
せっかちな人はこちら → https://yfujii08.shinyapps.io/pkpdsimulator/
一番出来のいいエクセルは → https://home.hiroshima-u.ac.jp/r-nacamura/
話は変わるのだが、手術の最中に鎮痛(痛み止め)としてフェンタニルという麻薬を使う。この薬は適当に投与していてもなんとかなるが、本当にいい感じに使いたかったら、痛みがとれる濃度に収めつつ、かつ、副作用として呼吸抑制(息をしてくれない)を起こさない濃度にしなければならない。
そして、手術の最中は適当に投与していてもいい(手術中は人工呼吸器という機械で息をしてくれているのは保証されているから)が、手術が終わったあとも、上記のいい感じの濃度に保って鎮痛を継続して図りたいのである。
ここで、濃度のシミュレーションアプリとして、FentaSim(無料)、iTIVA anesthesia(無料といいながらアプリ内課金でぜんぜん無料で使えない)、AnestAssist PK/PD(潔く有料)というものがいくつか出ているのだが、これらのアプリがやってくれているのは、結局、
・濃度シミュレーション
・UI として可視化
というわけである。濃度シミュレーションはAnesthesiology. 1990 Dec;73(6):1091-102.あたりから必要な微分方程式とパラメータを持ってきている。要は3コンパートメントモデルと効果部位を想定して、コンパートメント から
への移動を
と表記する。ここで効果部位は
, メイン(というか静脈注射されて薬が一気に流入してくるコンパートメントのことで、たいてい1番目)コンパートメントから他のコンパートメント以外への排泄を0 として、使われるパラメータは
,
,
,
,
,
となる。
というわけでフェンタニルのパラメータを使って開始時と1時間のときに適当に投与した結果がこちら。
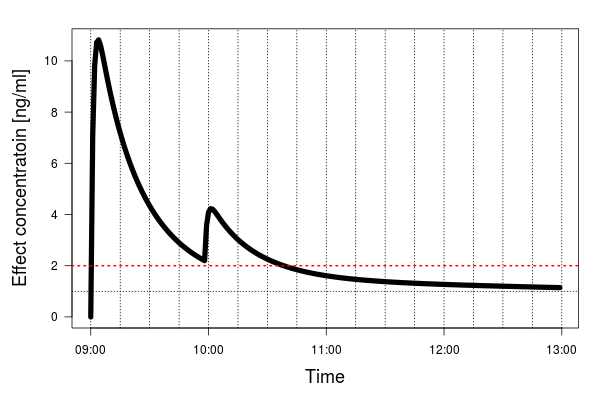
# patient setting weight <- 50 height <- 160 gender <- "M" LBM <- ifelse(gender == "M", 1.1*weight-128*(weight/height)^2,1.07*weight-148*(weight/height)^2) timestep <- 120 # parameter setting k_1N <- c(0.0827, 0.471, 0.225) k_N1 <- c(0.102, 0.006) k_e0 <- 0.114 k <- c(k_1N, k_N1, k_e0)/(60/timestep) names(k) <- c(sprintf("k1%d", c(0, 2, 3)), sprintf("k%d1", 2:3), "ke0") k1 <- k["k10"] + k["k12"] + k["k13"] V1 <- 0.105*weight CLs <- k[c("k10", "k12", "k13")]*(60/timestep)*V1 V2 <- CLs[2]/(k["k21"]*(60/timestep)) V3 <- CLs[3]/(k["k31"]*(60/timestep)) # 適当な投与 X[1, "Bolus"] <- 123000*(60/timestep) X[60, "Bolus"] <- 100000*(60/timestep) X <- rbind(0, X) for(i in 1:(nrow(X)-1)){ X$dA1_1[i+1] <- k["k21"]*X$A2[i]+k["k31"]*X$A3[i]-k1*X$A1[i]+X$Cont[i+1]/(60/timestep) X$dA2_1[i+1] <- k["k12"]*X$A1[i] - k["k21"]*X$A2[i] X$dA3_1[i+1] <- k["k13"]*X$A1[i] - k["k31"]*X$A3[i] X$dAe_1[i+1] <- k["ke0"]*(X$A1[i] - X$Ae[i]) X$dA1_2[i+1] <- k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)-k1*(X$A1[i]+X$dA1_1[i+1]/2)+X$Cont[i+1]/(60/timestep) X$dA2_2[i+1] <- k["k12"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2) X$dA3_2[i+1] <- k["k13"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2) X$dAe_2[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_1[i+1]/2 - (X$Ae[i]+X$dAe_1[i+1]/2)) X$dA1_3[i+1] <- k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)-k1*(X$A1[i]+X$dA1_2[i+1]/2)+X$Cont[i+1]/(60/timestep) X$dA2_3[i+1] <- k["k12"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2) X$dA3_3[i+1] <- k["k13"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2) X$dAe_3[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_2[i+1]/2 - (X$Ae[i]+X$dAe_2[i+1]/2)) X$dA1_4[i+1] <- k["k21"]*(X$A2[i]+X$dA2_3[i+1])+k["k31"]*(X$A3[i]+X$dA3_3[i+1])-k1*(X$A1[i]+X$dA1_3[i+1])+X$Cont[i+1]/(60/timestep) X$dA2_4[i+1] <- k["k12"]*(X$A1[i]+X$dA1_3[i+1]) - k["k21"]*(X$A2[i]+X$dA2_3[i+1]) X$dA3_4[i+1] <- k["k13"]*(X$A1[i]+X$dA1_3[i+1]) - k["k31"]*(X$A3[i]+X$dA3_3[i+1]) X$dAe_4[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_3[i+1] - (X$Ae[i]+X$dAe_3[i+1])) X$A1[i+1] <- X$A1[i]+(X$dA1_1[i+1]+2*X$dA1_2[i+1]+2*X$dA1_3[i+1]+X$dA1_4[i+1])/6+X$Bolus[i+1]/(60/timestep) X$A2[i+1] <- X$A2[i]+(X$dA2_1[i+1]+2*X$dA2_2[i+1]+2*X$dA2_3[i+1]+X$dA2_4[i+1])/6 X$A3[i+1] <- X$A3[i]+(X$dA3_1[i+1]+2*X$dA3_2[i+1]+2*X$dA3_3[i+1]+X$dA3_4[i+1])/6 X$Ae[i+1] <- X$Ae[i]+(X$dAe_1[i+1]+2*X$dAe_2[i+1]+2*X$dAe_3[i+1]+X$dAe_4[i+1])/6 X$Cp[i+1] <- X$A1[i+1]/V1/1000 X$Ce[i+1] <- X$Ae[i+1]/V1/1000 X$C1[i+1] <- X$A1[i+1]/V1/1000 X$C2[i+1] <- X$A2[i+1]/V2/1000 X$C3[i+1] <- X$A3[i+1]/V3/1000 } X <- X[-1,] t0 <- Sys.time() tx <- t0 + (seq(nrow(X))-1)*60 yl <- c(0, max(X$Ce)) par(mar=c(5, 5, 1, 1), las=1) plot(tx, X$Ce, type="l", ylim=yl)
さて、自分のPCが手元にあるときは自由にパラメータを設定して任意にシミュレーションできるが、PCがないときやRを使わない他の人に使ってもらいたいときに困る。このようなときにアプリ化しておくと便利である。これはShiny でできる。
Shiny のサンプルはRstudio の新規作成 > shiny にすれば、ヒストグラムを描いてbin をインタラクティブに操作できるものが作成される。出力はoutput$hoge に入っていて、サンプルではoutput$distPlot でヒストグラムが翔ようになっている。ここをいじる。
output にはinput$bins から必要な変数が取得できるので、ここをいじる。生値を入力するのか、スライドバーで設定するのか、選択肢を作るのかはhogehogeInput 関数で設定できる。
アプリを作るにはui.R とapp.R を作る必要があるようだが、app.R 内にui とserver をごちゃごちゃ書きこめばなんとかなるっぽい。アプリを公開するには手っ取り早いのはshinyserver を使う。rmd をRpubs に公開するのと同じように、Rstudio から run app したあとに出力されるhtml の右上に公開、というボタンがあるのでそれを押すと、「アカウントある?」と聞かれるのでとりあえずアカウントを作る。アカウントを作成したあとは、アプリ公開のためのキーを取得して紐づけする必要があるので、SECRET を直して他人に見られないようにRにコピペする。これはツイッターAPI などを使う人はやったことがあるはず。
そのあと、Rstudio から公開しようとするとなぜかできなかった。なのでRコンソールから
rsconnect::deployApp('path/to/your/app')
とするとなぜかうまくいった。このとき、app はapp.R ではなく、app.R があるディレクトリを参照する。
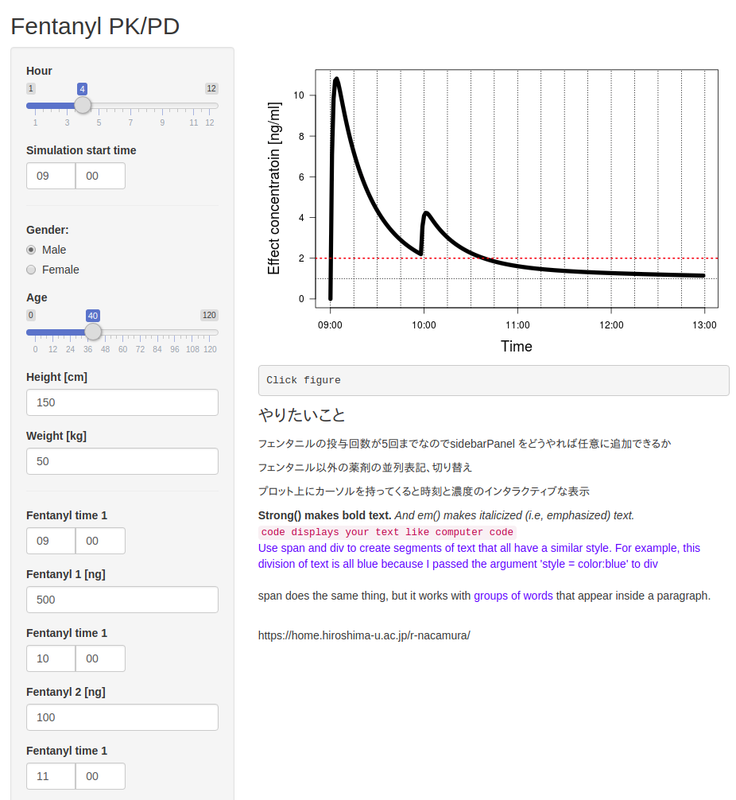
### app.R # # This is a Shiny web application. You can run the application by clicking # the 'Run App' button above. # # Find out more about building applications with Shiny here: # # http://shiny.rstudio.com/ # # http://shiny.rstudio.com/gallery/ # https://shiny.qhmqk.me/shinywidgets/ library(shiny) library(shinyTime) # rsconnect::deployApp('path/to/your/app') # Define UI for application that draws a histogram ui <- fluidPage( # Application title titlePanel("Fentanyl PK/PD"), # Sidebar with a slider input for number of bins sidebarLayout( sidebarPanel( sliderInput("hour", "Hour", min = 1, max = 12, value = 4, step=1), #dateInput("date", label = h3("Date input"), value = Sys.Date()), timeInput("starttime", label = "Simulation start time", value = strptime("09:00:00", "%T"), seconds=FALSE), hr(), radioButtons("gender", "Gender:", c("Male" = "M", "Female" = "F")), sliderInput("age", label="Age", value = 40, min = 0, max = 120, step=1), numericInput("height", label = "Height [cm]", min=1, max=250, value = 150), numericInput("weight", label = "Weight [kg]", min=1, max=150, value = 50), hr(), timeInput("ftime1", label = "Fentanyl time 1", value = strptime("09:00:00", "%T"), seconds=FALSE), numericInput("iv1", label = "Fentanyl 1 [ng]", min=0, max=1000, value = 50), timeInput("ftime2", label = "Fentanyl time 2", value = strptime("10:00:00", "%T"), seconds=FALSE), numericInput("iv2", label = "Fentanyl 2 [ng]", min=0, max=1000, value = 0), timeInput("ftime3", label = "Fentanyl time 3", value = strptime("11:00:00", "%T"), seconds=FALSE), numericInput("iv3", label = "Fentanyl 3 [ng]", min=0, max=1000, value = 0), timeInput("ftime4", label = "Fentanyl time 4", value = strptime("12:00:00", "%T"), seconds=FALSE), numericInput("iv4", label = "Fentanyl 4 [ng]", min=0, max=1000, value = 0), timeInput("ftime5", label = "Fentanyl time 5", value = strptime("13:00:00", "%T"), seconds=FALSE), numericInput("iv5", label = "Fentanyl 5 [ng]", min=0, max=1000, value = 0), hr(), #numericInput("ylim", label = h3("ylim [ng/ml]"), min=0, max=50, value=3), sliderInput("ylim", label="ylim [ng/ml]", value = 0, min = 0, max = 5, step=0.5), sliderInput("Cont_conc", label="ivPCA [ng/hr]", value = 0, min = 0, max = 50, step=5), timeInput("ivPCAstart", label = "ivPCA start time", value = strptime("12:00:00", "%T"), seconds=FALSE), hr(), fluidRow(column(3, verbatimTextOutput("value"))) #submitButton("Submit") ), # Show a plot of the generated distribution mainPanel( plotOutput("distPlot", click="plot_click"), verbatimTextOutput("info"), p("やりたいこと", style = "font-family: 'times'; font-size: 16pt"), p("フェンタニルの投与回数が5回までなのでsidebarPanel をどうやれば任意に追加できるか"), p("フェンタニル以外の薬剤の並列表記、切り替え"), p("プロット上にカーソルを持ってくると時刻と濃度のインタラクティブな表示"), strong("Strong() makes bold text."), em("And em() makes italicized (i.e, emphasized) text."), br(), code("code displays your text like computer code"), div("Use span and div to create segments of text that all have a similar style. For example, this division of text is all blue because I passed the argument 'style = color:blue' to div", style = "color:blue"), br(), p("span does the same thing, but it works with", span("groups of words", style = "color:blue"), "that appear inside a paragraph."), br(), p("https://home.hiroshima-u.ac.jp/r-nacamura/") ) ) ) # Define server logic required to draw a histogram server <- function(input, output) { test <- 1 output$distPlot <- renderPlot({ # generate bins based on input$bins from ui.R weight <- input$weight comp <- c(1:3, "e") cname <- c("Bolus", "Cont", sprintf("A%s", comp), mapply(function(z) sprintf("dA%s_%d", comp, z), 1:4), "Cp", sprintf("C%s", comp)) X <- matrix(0, input$hour*60, length(cname), dimnames=list(NULL, cname)) X <- as.data.frame(X) timestep <- 60 k_1N <- c(0.0827, 0.471, 0.225) k_N1 <- c(0.102, 0.006) k_e0 <- 0.114 k <- c(k_1N, k_N1, k_e0)/(60/timestep) names(k) <- c(sprintf("k1%d", c(0, 2, 3)), sprintf("k%d1", 2:3), "ke0") k1 <- k["k10"] + k["k12"] + k["k13"] V1 <- 0.105*weight CLs <- k[c("k10", "k12", "k13")]*(60/timestep)*V1 V2 <- CLs[2]/(k["k21"]*(60/timestep)) V3 <- CLs[3]/(k["k31"]*(60/timestep)) t0 <- input$starttime - 61*0 tx <- t0 + (seq(nrow(X))-1)*60 X[tx > input$ivPCAstart, "Cont"] <- input$Cont_conc/60*1000 X <- na.omit(X) #X[1, "Bolus"] <- input$iv1*1000*(60/timestep) #X[100, "Bolus"] <- 150000*(60/timestep) X <- rbind(0, X) ### together #ivs <- unlist(mapply(function(z) eval(parse(text=z)), sprintf("input$iv%d", 1:5))) #ft <- unlist(mapply(function(z) eval(parse(text=z)), sprintf("input$ftime%d", 1:5))) #idx <- mapply(function(z) tail(which(tx < z), 1), ft) #X[idx, "Bolus"] <- ivs*(60/timestep) ### each X[tail(which(tx < input$ftime1+61), 1), "Bolus"] <- input$iv1*1000*(60/timestep) X[tail(which(tx < input$ftime2), 1), "Bolus"] <- input$iv2*1000*(60/timestep) X[tail(which(tx < input$ftime3), 1), "Bolus"] <- input$iv3*1000*(60/timestep) X[tail(which(tx < input$ftime4), 1), "Bolus"] <- input$iv4*1000*(60/timestep) X[tail(which(tx < input$ftime5), 1), "Bolus"] <- input$iv5*1000*(60/timestep) for(i in 1:(nrow(X)-1)){ X$dA1_1[i+1] <- k["k21"]*X$A2[i]+k["k31"]*X$A3[i]-k1*X$A1[i]+X$Cont[i+1]/(60/timestep) X$dA2_1[i+1] <- k["k12"]*X$A1[i] - k["k21"]*X$A2[i] X$dA3_1[i+1] <- k["k13"]*X$A1[i] - k["k31"]*X$A3[i] X$dAe_1[i+1] <- k["ke0"]*(X$A1[i] - X$Ae[i]) X$dA1_2[i+1] <- k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)-k1*(X$A1[i]+X$dA1_1[i+1]/2)+X$Cont[i+1]/(60/timestep) X$dA2_2[i+1] <- k["k12"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2) X$dA3_2[i+1] <- k["k13"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2) X$dAe_2[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_1[i+1]/2 - (X$Ae[i]+X$dAe_1[i+1]/2)) X$dA1_3[i+1] <- k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)-k1*(X$A1[i]+X$dA1_2[i+1]/2)+X$Cont[i+1]/(60/timestep) X$dA2_3[i+1] <- k["k12"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2) X$dA3_3[i+1] <- k["k13"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2) X$dAe_3[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_2[i+1]/2 - (X$Ae[i]+X$dAe_2[i+1]/2)) X$dA1_4[i+1] <- k["k21"]*(X$A2[i]+X$dA2_3[i+1])+k["k31"]*(X$A3[i]+X$dA3_3[i+1])-k1*(X$A1[i]+X$dA1_3[i+1])+X$Cont[i+1]/(60/timestep) X$dA2_4[i+1] <- k["k12"]*(X$A1[i]+X$dA1_3[i+1]) - k["k21"]*(X$A2[i]+X$dA2_3[i+1]) X$dA3_4[i+1] <- k["k13"]*(X$A1[i]+X$dA1_3[i+1]) - k["k31"]*(X$A3[i]+X$dA3_3[i+1]) X$dAe_4[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_3[i+1] - (X$Ae[i]+X$dAe_3[i+1])) X$A1[i+1] <- X$A1[i]+(X$dA1_1[i+1]+2*X$dA1_2[i+1]+2*X$dA1_3[i+1]+X$dA1_4[i+1])/6+X$Bolus[i+1]/(60/timestep) X$A2[i+1] <- X$A2[i]+(X$dA2_1[i+1]+2*X$dA2_2[i+1]+2*X$dA2_3[i+1]+X$dA2_4[i+1])/6 X$A3[i+1] <- X$A3[i]+(X$dA3_1[i+1]+2*X$dA3_2[i+1]+2*X$dA3_3[i+1]+X$dA3_4[i+1])/6 X$Ae[i+1] <- X$Ae[i]+(X$dAe_1[i+1]+2*X$dAe_2[i+1]+2*X$dAe_3[i+1]+X$dAe_4[i+1])/6 X$Cp[i+1] <- X$A1[i+1]/V1/1000 X$Ce[i+1] <- X$Ae[i+1]/V1/1000 X$C1[i+1] <- X$A1[i+1]/V1/1000 X$C2[i+1] <- X$A2[i+1]/V2/1000 X$C3[i+1] <- X$A3[i+1]/V3/1000 } X <- X[-1,] yl <- c(0, ifelse(input$ylim==0, max(X$Ce), ifelse(input$ylim<max(X$Ce), input$ylim, max(X$Ce)))) par(mar=c(5, 5, 2, 1), las=1, cex.lab=1.5) plot(tx, X$Ce, type="n", ylim=yl, xlab="Time", ylab="Effect concentratoin [ng/ml]") lines(tx, X$Ce, lwd=7) abline(h=1.0, lty=3) abline(h=2.0, lty=3, col="red", lwd=2) abline(v=seq.POSIXt(tx[1], tail(tx, 1)+100, by=15*60), lty=3) if(input$Cont_conc > 0){ x_ivPCA <- as.numeric(input$ivPCAstart) abline(v=x_ivPCA, lty=3, col="yellow") text(x_ivPCA, par()$usr[4], "ivPCA", pos=3, xpd=TRUE, cex=1.6) } }) output$info <- renderText({ xy_str <- function(e) { if(is.null(e)) return("Click figure\n") click_time <- format(as.POSIXlt(as.numeric(e$x), origin="1970-01-01"), "%H:%M") sprintf("Ce is %.4f at %s", e$y, click_time) #paste0("x=", round(e$x, 1), " y=", round(e$y, 1), "\n") } paste0( xy_str(input$plot_click) ) }) } # Run the application shinyApp(ui = ui, server = server)
