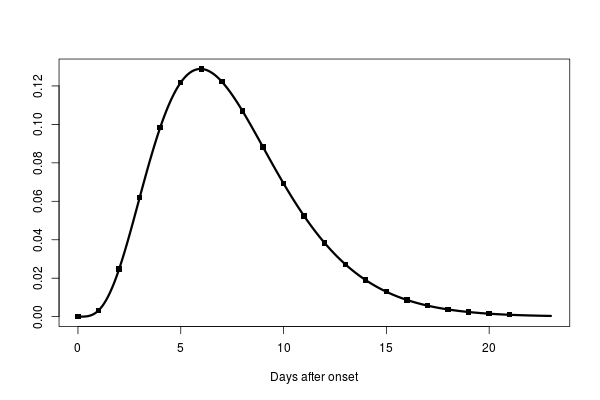
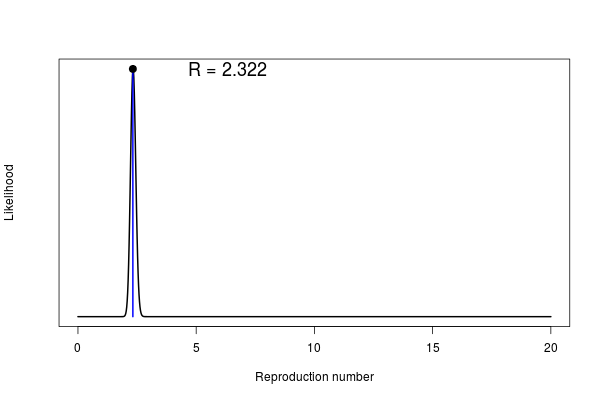
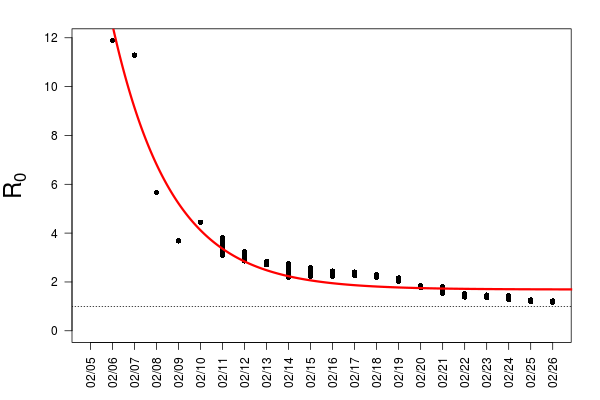
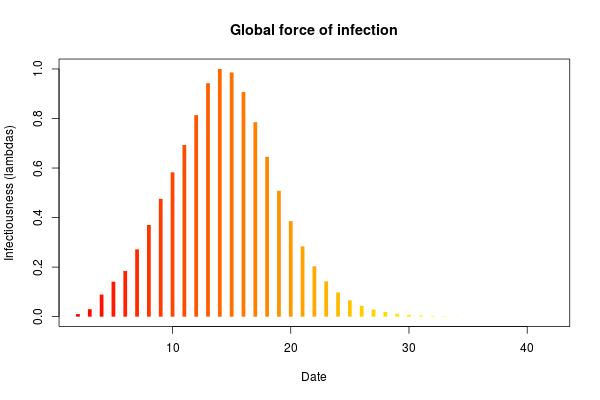
これに、打ち切りデータがあるときの平均値の推定問題がある。

StanとRでベイズ統計モデリング (Wonderful R)
- 作者:健太郎, 松浦
- 発売日: 2016/10/25
- メディア: 単行本
<25となっているので、このデータを無視して平均値を推定すると、本当にデータがあったときより過大評価するだろう、ということ。打ち切りの場合に
<25を無視するのではなく、<25となったときにオリジナルのコードは
data { int N_obs; int N_cens; real Y_obs[N_obs]; real L; } parameters{ real<lower=0> m; real<lower=0> s_Y; } model{ for(n in 1:N_obs){ Y_obs[n] ~ normal(m, s_Y); } target += N_cens * normal_lcdf(L | m, s_Y); }
となっているが、打ち切りのデータにインデックスをつけてforを回すパターンにした。また、サンプリングはY ~ normal() でもよいが、target 記法で合わせてみようと思って
サンプリングの部分はtarget += _lpdf(data | parameters)(そのデータをサンプリングする確率密度lpdf)
打ち切りの部分はtarget += _lcdf(censor | parameters)( までの累積確率分布
lcdf)
となる。
rstan で推定した、正規分布のパラメータ の事後分布である。この事後分布の最頻値は、シミュレーションで設定した真の値 50 より(たまたま)小さい。
は40 に設定したが、
を下回った値は
<40 と記録されてしまうとして、このデータを除外して平均を推定した場合はOmit の縦線となり、Censor の縦線より大きくなっている。

さて、rstanで打ち切りデータを考慮した場合と、除外して平均を推定した場合とで、どれくらい推定値が変わるかをシミュレーションした。
除外した場合は、真の値よりほとんどの場合で大きい値を推定してしまう。
打ち切りを考慮した場合は、だいたい真の値を推定しているようだった。

library(rstan) rstan_options(auto_write=TRUE) options(mc.cores=parallel::detectCores()) # 偏差値っぽい感じで m_true <- 50 s_true <- 10 n <- 30 y <- rnorm(n, m_true, s_true) L <- 40 idx <- which(y < L) y0 <- replace(y, idx, L) code <- " data { int N; real Y[N]; int I[N]; real L; } parameters{ real<lower=0> m; real<lower=0> s; } model{ for(i in 1:N){ if(I[i] == 0){ // データがある場合 target += normal_lpdf(Y[i] | m, s); } else { // 打ち切りの場合 target += normal_lcdf(L | m, s); } } } " m0 <- stan_model(model_code=code) standata <- list(N=length(y), Y=y, I=replace(rep(0, length(y)), y < L, 1), L=L) fit <- sampling(m0, standata, iter=1000, warmup=300, chain=4) ex <- extract(fit, pars=head(fit@model_pars, -1)) library(vioplot) par(mar=c(3, 2, 3, 2)) plot(y, rep(0.99, n), pch=16, col=2, xlab="", ylab="", ylim=c(0.95, 1.5), yaxt="n") vioplot(ex$m, horizontal=TRUE, side="right", ylim=range(y), add=TRUE) pa <- par()$usr abline(v=m_true); text(m_true, pa[4], "True", xpd=TRUE, srt=0, pos=3) abline(v=median(ex$m)); text(median(ex$m), pa[4], "Censor", xpd=TRUE, srt=0, pos=3, offset=1.5) abline(v=mean(y[y>L])); text(mean(y[y>L]), pa[4], "Omit", xpd=TRUE, srt=0, pos=3) abline(v=L); text(L, pa[4], "L", xpd=TRUE, srt=0, pos=3) # 除外した平均と、rstanで推定した平均の違いをシミュレーション iter <- 1000 M <- matrix(0, iter, 2) for(i in 1:iter){ y <- rnorm(n, m_true, s_true) standata <- list(N=length(y), Y=y, I=replace(rep(0, length(y)), y < L, 1), L=L) fit <- sampling(m0, standata, iter=1000, warmup=300, chain=4) ex <- extract(fit, pars=head(fit@model_pars, -1)) M[i, ] <- c(mean(y[y > L]), median(ex$m)) } xylim <- range(M) par(mar=c(5, 5, 3, 3), cex.lab=1.5, cex.axis=1.2) plot(M, xlim=xylim, ylim=xylim, xlab="L 以下を除外した平均", ylab="累積確率を考慮した平均", pch=16) abline(v=m_true, h=m_true, lty=3, col=4) abline(0, 1, lty=3, col=4) vioplot(M[,1], at=par()$usr[4], add=TRUE, side="right", xpd=TRUE, horizontal=TRUE, wex=3) vioplot(M[,2], at=par()$usr[2], add=TRUE, side="right", xpd=TRUE, wex=3)