GF(仮)のキャラがニーソかどうか知りたかったわけだが、興味深いコメントをもらった。
たぶんこれは正しいと思う。というのも、サンプルが小さいのにデータベクトルがでかすぎる。
コメントのランダム分類は意味不明(ゴメンナサイ)なので、真面目に適当に分類した時(日本語おかしい)の判定性能をシミュレーションで確かめてみる。
交差検証用に14に分割したとき、112のサンプルは学習用データ104, 検証用データ8に分かれる。
ここで、学習用データのニーソフラグの分布から、ニーソである確率が出るので、検証用データをその確率に従って適当に0/1のサンプリングをする。
ちなみにニーソは20%程度なので、1が20%の確率でサンプリングされる。
適当な回数繰り返して真の値に収束する感じか確かめる。
結果
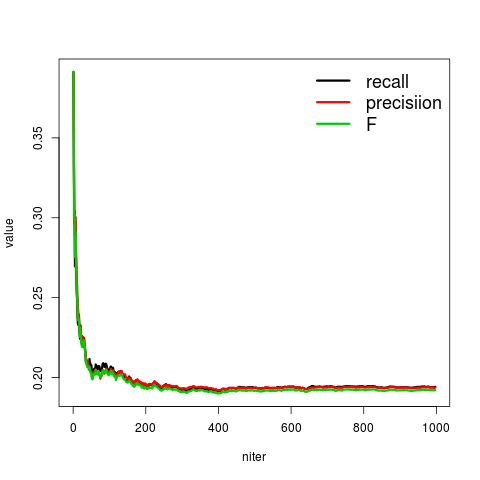
mean lower upper recall 0.19414766 -0.0004093074 0.0004093074 precisiion 0.19364392 -0.0003204273 0.0003204273 F 0.19211794 -0.0003345967 0.0003345967 MMC -0.01434838 -0.0004917381 0.0004917381
適当に分類してもF値0.19はある。
知 っ て た
niter <- 1000 # シミュレーション回数 res <- matrix(0, nr=niter, nc=4) # 格納 for(k in seq(niter)){ vec <- rep(-1, length(knee)) for(j in seq(ncol(cv_group))){ p_true <- mean(knee[cv_group[, -j]]) # 学習用データのなかでのニーソの分布確率 vec[cv_group[, j]] <- sample(0:1, size=nrow(cv_group), prob=c(1-p_true, p_true), replace=TRUE) } res[k, ] <- unlist(stats(table(knee, vec))) } colnames(res) <- names(stats(table(knee, vec))) res <- res[!apply(res == 0, 1, any), ] # F値が計算できない場合もあるので削除 # 信頼区間 ci <- mapply(function(x) qt(c(0.025, 0.975), nrow(res))*var(res[, x])/sqrt(nrow(res)), seq(ncol(res))) m <- t(rbind(colMeans(res), ci)) colnames(m) <- c("mean", "lower", "upper") # 収束の様子をプロットする matplot(apply(res[, -4], 2, cumsum)/seq(nrow(res)), type="l", lty=1, lwd=3, xlab="niter", ylab="value") legend("topright", legend=colnames(res)[-4], lty=1, lwd=3, bty="n", col=seq(3), cex=1.5)