時系列データにt 検定を行うことに関して、すごいもにょっていたのだが、そもそもstan 神が既にモデル化してくれていた。
リンクでは2階差分と、変化点検出のコーシー分布の合わせ技を用いている。
そのままパクってやってみる。
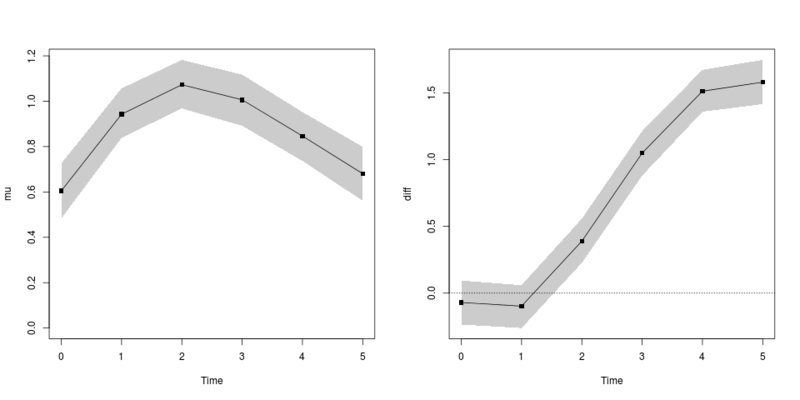
diの95%ベイズ信頼区間が0を含んでいない期間が差がある期間と言えるでしょう。さらに、どこから差がありそうなのか、どれほど差がありそうなのかも確率付きで述べることができます。
ということが、stan による柔軟なモデリングで述べることができます。
話は飛ぶけど、読んだ。

はじめての 統計データ分析 ―ベイズ的〈ポストp値時代〉の統計学―
- 作者: 豊田秀樹
- 出版社/メーカー: 朝倉書店
- 発売日: 2016/06/02
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (11件) を見る
書評はまた書くけれども、この時系列データと同じように、この本では、例えば分散分析や分割用を用いて、「どことどこが比較して差があるか」という、実際のデータ解析でも気になる点を実例を用いて解説してくれている。
古典的なp値検定では、この問に答えることはクッソ難しかったが、ベイズだとかなり柔軟に対応できるようになるので、いいと思います。
# berobero.stan data { int N_time; int N0; int N1; vector[N_time] Y0[N0]; vector[N_time] Y1[N1]; } parameters { vector[N_time] mu; real di0; vector<lower=-pi()/2, upper=pi()/2>[N_time-1] di_unif; real<lower=0> s_mu; real<lower=0> s_di; real<lower=0> s_Y; } transformed parameters { vector[N_time] di; vector[N_time] y1_mean; di[1] <- di0; for (t in 2:N_time) di[t] <- di[t-1] + s_di*tan(di_unif[t-1]); y1_mean <- mu + di; } model { for (t in 3:N_time) mu[t] ~ normal(2*mu[t-1] - mu[t-2], s_mu); for (n in 1:N0) Y0[n] ~ normal(mu, s_Y); for (n in 1:N1) Y1[n] ~ normal(y1_mean, s_Y); }
# データ作り x <- seq(0, 10, length=100) d <- 0:5 n <- 10 b <- 0.5 g <- exp(-1/2.5)/exp(-1) f0 <- function(x) ((x)^2)*exp(-x/1) + b f1 <- function(x) ((x)^2)*exp(-x/2.5)/g + b y0 <- replicate(n, f0(d) + rnorm(length(d), sd=0.2)) y1 <- replicate(n, f1(d) + rnorm(length(d), sd=0.2)) y01 <- list(y0, y1) library(rstan) standata <- list(N_time=length(d), N0=ncol(y01[[1]]), N1=ncol(y01[[2]]), Y0=t(y01[[1]]), Y1=t(y01[[2]])) stanmodel01 <- stan_model("berobero.stan") fit0 <- sampling(stanmodel01, data=standata, chains=3, warmup=1000, iter=3000, seed=1234) ex0 <- extract(fit0) alpha <- 0.05 Y_ci <- t(apply(ex0$mu, 2, quantile, c(alpha/2, 1-alpha/2))) Y_me <- apply(ex0$mu, 2, median) yl <- range(0, Y_ci) xp <- c(d, rev(d)) yp <- c(Y_ci[,1], rev(Y_ci[,2])) matplot(d, Y_ci, type="n", xlab="Time", ylab="mu", ylim=yl) polygon(xp, yp, col=grey(0.8), border=NA) lines(d, Y_me, type="o", pch=15) Y_ci <- t(apply(ex0$di, 2, quantile, c(alpha/2, 1-alpha/2))) Y_me <- apply(ex0$di, 2, median) yl <- range(0, Y_ci) xp <- c(d, rev(d)) yp <- c(Y_ci[,1], rev(Y_ci[,2])) matplot(d, Y_ci, type="n", xlab="Time", ylab="diff", ylim=yl) polygon(xp, yp, col=grey(0.8), border=NA) lines(d, Y_me, type="o", pch=15) abline(h=0, lty=3)
共通のベースライン(mu;左図)と、変異体と野生型の差(di;右図)の結果。
時刻0, 1 の時点では差が出ないようにデータを作っていて、2 以降から差が出るようになっている。
また、シミュレーションの曲線では、緑の曲線が時刻 5 で頭打ちになるようにしていて、これもそれとなく推定されている。
個人的にはggplot2 が慣れていないから、これを描くスクリプトも載せてほしいな!!