
読んだ。
Bioinformatics(2009)25(22):2906-2912.
コピーナンバー(CNV)、発現データ(mRNA)、メチル化などのオミックスデータで、症例数 に対してパラメータ数
のデータ行列が複数ある。
各々の実験を勝手にやるのは、それはそれでいいが、CNV、発現、メチル化、プロテオームetc と 種類のいろいろなデータを取ったら、統合して考えたいと思うのは自然な発想である。
ここで、 次元のデータ行列
が、適当な係数行列
、latent と呼ばれる、裏で共通して存在しているであろう(だが、観測はできない)因子
と誤差
を用いて
として を求めたい。
iCluster パッケージにある。
あるオミックス実験(gene exprssion とか)は の行列データをヒートマップ化して、階層的クラスタリングで系統樹を書くことが多いが、
個のオミックス実験で
症例たちがk-means で
クラスターに分類できる、とする。
は平均で中央揃えした
次元の行列である。
は
群でわけられ(
)、クラスター平均ベクトル
について郡内誤差を最小化する。
となる行列を作るが、ここで
は
番目のクラスター内のサンプル数で、
であり、 の部分は
個ある。また、
である。
をグラム行列(
のとき、内積[tex:G_{ij}=
と表されるらしく、前者はtotal variance, 後者はbetween-cluster varianvce となるので、後者を最大化すれば全体の最小化が達成される。
は連続量でもいいので、
個の降順のeigenvalue で
とできる。
大事なのはGaussian latent variable である で
を分解する。
はデータ行列で
、
は係数行列で
、
はlatent variable で
、
は対角成分が共分散の行列で
である。
EM アルゴリズムでなんやかんやしてたら解ける。また、 問題のときにスパースに解くことができるし、LASSO による係数選択も行う。
が大きくなってスパースになると、
の多くが0 になる。
は係数行列なので、遺伝子発現やCNV など各遺伝子(
なのでこれはたいていすごい大きな数である)の作用の強さを決めている。大きい
になるとほとんどが0 になり、本当に作用の強い遺伝子(
)しか残らなくなるので、解釈は楽になる。
iClusterPlus
適切なk-means クラスターの数はproportion of deeviance (POD) という統計量で測ることができ、shrinkage parameter のときのPOD が最小化のときが最もよさそうなクラスターの分け方になる。論文では
である。
par(mfrow=c(1, 2)) for(i in 1:2){ heatmap.2(t(breast.chr17[[i]]), trace="none", col=gplots::greenred(100), scale="row", hclustfun=function(x) hclust(x, method="ward.D2")) }
CNV. Fig2A に相当する。
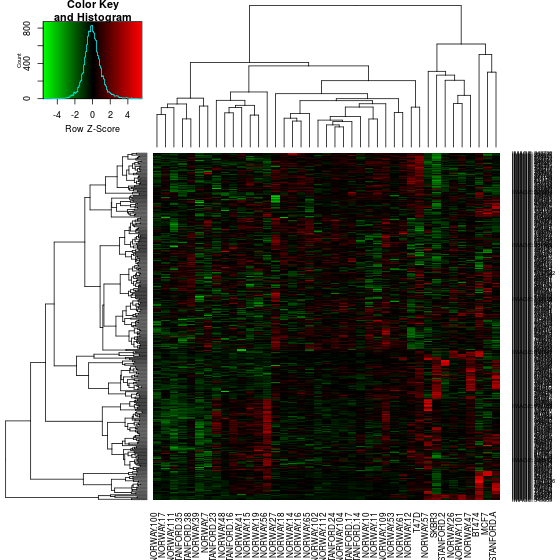
gene expression. Fig2A に相当する。

library(iCluster) data(breast.chr17) fit <- mapply(function(k) iCluster(breast.chr17, k=k, lambda=c(0.2,0.2)), 2:5, SIMPLIFY=FALSE) par(mfrow=c(2, 2)) for(i in seq(fit)) plotiCluster(fit=fit[[i]], label=rownames(breast.chr17[[2]])) pod <- mapply(compute.pod, fit)
plotHeatmap(fit[[which.min(pod)]], datasets=breast.chr17)
k クラスターに分けたあとの、 個の実験のクラスタリング。行はパラメータ
, 列が症例
に対応している。

こうして新規に分けられたkクラスターで、生存曲線を書いて差があると、オミックス統合した解析がいいでしょう、となる。
論文では、乳癌のHER/ERBB, 肺癌のEGFR などをメルクマールに解析している。
