RでCTっぽいことをしようと思ったのだが、3D画像がよくわからない。ということで昔Rで年賀状という記事を見たことがあったので、これよりThe Stanford 3D Scanning Repositoryというところに行ってブッダの治療をすることにしよう。
happy_recon.tar.gz というファイルを取ってきて、コンソールで
egrep '^[0-9.\-][0-9.\-]* [0-9.\-][0-9.\-]* [0-9.\-][0-9.\-]* $' ~/Desktop/happy_recon/happy_vrip_res.ply > ~/Desktop/test.txt
とすると、ヘッダー情報を除いてxyzの座標データを取れる。
buddha <-matrix(scan(file="~/Desktop/test.txt"),ncol=3,byrow=T) xyzl <- apply(buddha, 2, range) library(rgl) #ブッダは縦に長いのでアスペクト比を変更する plot3d(buddha[order(buddha[, 2]), ], aspect=abs(apply(xyzl, 2, diff)), col=rainbow(nrow(buddha)), xlim=xyzl[, 1], ylim=xyzl[, 2], zlim=xyzl[, 3])
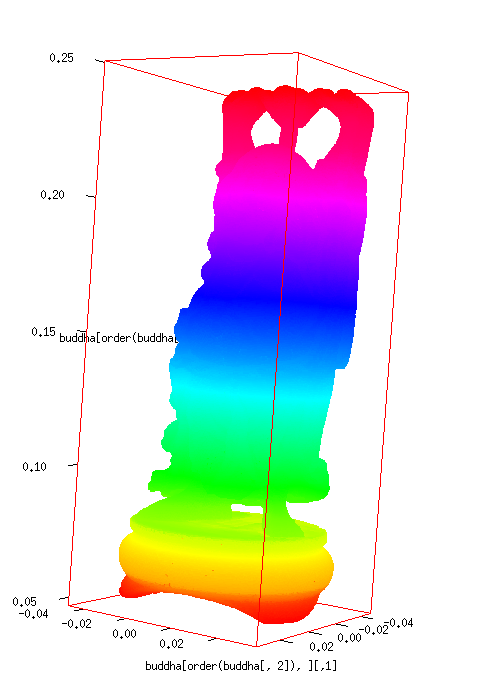
座標はブッダの体表面だけなので、体内に腫瘍っぽいものを作ろう。
x0 <- 0; y0 <- 0.15; z0 <- 0 #癌の中心座標 y0_up <- 0.008 #腫瘍の最大y y0_dw <- 0.008 #腫瘍の最小y tn <- 100 y1 <- seq(y0+y0_up, y0-y0_dw, length=tn) theta <- seq(0, 2, length=100)*pi y2 <- rep(y1, each=length(theta)) s0 <- rep(1 - c(rev(seq(tn)), seq(tn)) / tn, each=tn/2) ra <- 0.005 tumor <- mapply(rep, rep(0, 3), length(y2)) tumor[, 2] <- y2 for(i in seq(tn-1)){ for(j in seq(theta)){ tumor[tn*(i-1) + j, c(1, 3)] <- c(cos(theta[j]), sin(theta[j])) * ra * s0[tn*(i-1) + j] }} y0 <- 90000 plot3d(buddha[order(buddha[, 2])[1:y0], ], aspect=abs(apply(xyzl, 2, diff)), col=rainbow(nrow(buddha)), xlim=xyzl[, 1], ylim=xyzl[, 2], zlim=xyzl[, 3]) points3d(tumor)

あとはこれに放射線を当てるわけだが、三次元はきついぞ…
なので二次元で逃げる。