変化点検出をしたいしたいといって、ついにやることに。
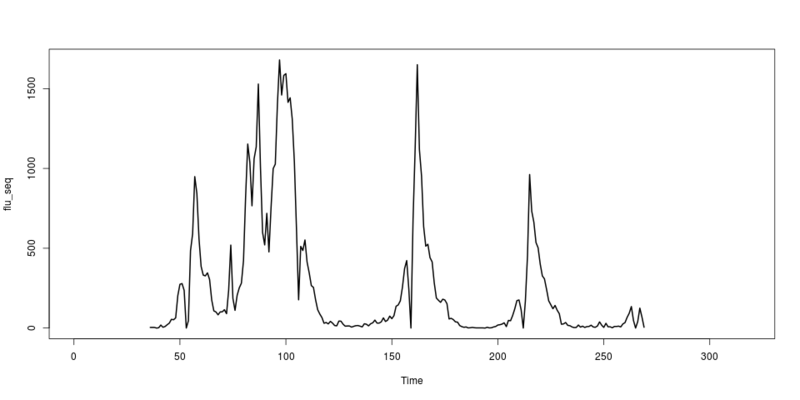
国立感染症研究所からインフルエンザ定点観測データを入手して、流行予測をしよう。
変化点検出パッケージChangeAnomalyDetectionを使ってみる。
library(ChangeAnomalyDetection) #データは下に flu <- read.delim("flu_japan_2008_2013.txt", header=FALSE) week <- rep(seq(ncol(flu)), nrow(flu)) #週 flu_seq <- c(t(flu[rev(seq(nrow(flu))), ])) #ベクトルにする ts.plot(flu_seq) #時系列プロット
flu_cad <- c(na.omit(flu_seq)) week_na <- attributes(na.omit(flu_seq))
用意ができたのでよしやるぞ…
changeAnomalyDetection(flu_cad) 以下にエラー arima(train, order = order, ...) : non-stationary AR part from CSS
ファッ!?
関数の中身をみてみると、自己回帰移動平均(ARMA; AutoRegressive Moving Average model)というモデルのところでエラーになっているらしい。
ということでやめた!!すまん。
他に探すと、ベイズ変化点検出というbcpパッケージがあったので、これでやってみる。
library(bcp) flu_bcp <- bcp(flu_cad, w0 = 0.2, p0 = 0.2) plot(flu_bcp)
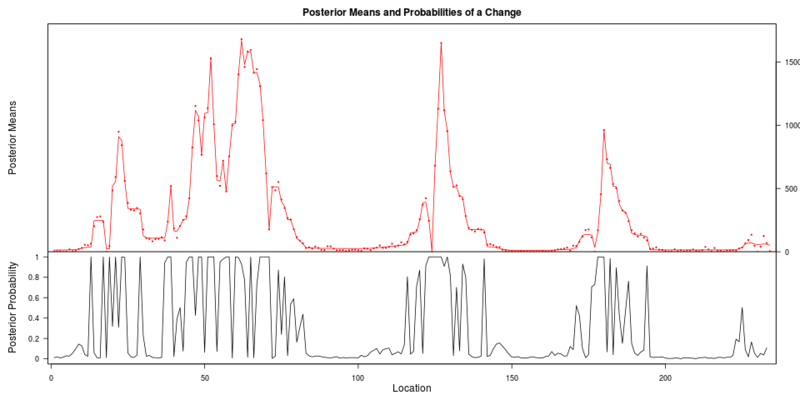
たぶん事後確率が高いとそうなんだろうけど、多すぎて「ここから流行開始です」って言えなさそう。
2008年36週目から2013年4週目までのインフルエンザ定点観測データ
40 125 69 5 170 455 962 732 663 536 503 401 327 308 242 171 146 121 142 112 90 24 26 35 17 15 7 3 4 19 6 12 4 9 10 18 7 5 13 39 18 4 30 8 8 2 10 10 12 7 26 34 67 93 135 48 0 681 1130 1650 1118 953 636 513 525 441 415 283 188 174 161 181 176 153 57 61 53 39 37 16 9 5 7 1 2 4 2 1 1 1 1 0 5 1 2 7 10 19 21 25 33 9 48 46 79 121 172 176 114 0 512 486 552 413 346 266 255 178 115 88 66 30 35 26 42 31 16 14 44 43 24 12 13 13 6 11 15 16 12 7 27 24 14 27 33 50 31 31 39 64 41 50 75 59 78 137 145 171 257 372 423 245 0 46 485 591 949 843 560 386 332 327 346 304 176 108 101 83 101 102 115 90 238 520 186 111 205 252 281 423 824 1153 1038 766 1061 1135 1529 1008 599 521 719 477 755 999 1027 1402 1680 1460 1583 1594 1414 1443 1309 1040 619 177 4 4 4 0 2 19 5 10 21 31 55 52 64 203 274 279 236 0