
マイクロアレイの解析っぽいことをやったのだが、KEGGによるパスウェイ解析やジーンオントロジー解析までは至らなかったので、これを参考にしながらやってみる。
Bioconductorを使った講義も予定されているので、そのときにはドヤ顔できるようにしよう。
データセットはGSE11324を使用する(Nat Genet. 2006 Nov;38(11):1289-97)。CELファイルがまとめられたRARファイルをダウンロードし、RARの解凍およびGZファイルの解凍をしておく。
実験デザインを確認しておくと、platformはGPL570 [HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array, 4点の観測時刻*3サンプルの12サンプルが収められている。
platformはChipManufacturerの中から、hgu133plus2.dbを確認する。
作業ディレクトリに飛んで、ディレクトリ内のすべての.CELファイルを読み込んで開始する。
setwd("/GSE11324/GSE11324_RAW/") # こんな感じのディレクトリができる source("http://bioconductor.org/biocLite.R") biocLite("hgu133plus2.db") # NCI60 で使用したアノテーション library(affy) data0 <- ReadAffy() data0
データの補正を行う。補正方法は死ぬほどあるらしい。とりあえずRMAを使うことにする。
eset <- rma(data0) # その他の補正法 eset <- expresso(data0, bgcorrect.method="none", normalize.method="quantiles", pmcorrect.method="pmonly", summary.method="dfw")#DFW法を実行し、結果をesetに保存。 eset <- expresso(data0, normalize.method="invariantset", bg.correct=FALSE, pmcorrect.method="pmonly", summary.method="liwong")#dChip(PM onlyモデル)を実行し、結果をesetに保存 eset <- expresso(data0, normalize.method="invariantset", bg.correct=FALSE, pmcorrect.method="subtractmm", summary.method="liwong")#dChip(PM-MMモデル)を実行し、結果をesetに保存 eset <- q.farms(data0) eset <- l.farms(data0) eset <- mmgmos(data0) eset <- gcrma(data0) eset <- justPlier(data0, normalize=TRUE) eset <- vsnrma(data0) eset <- mas5(data0) express <- exprs(eset) colnames(express) <- rep(c(0, 3, 6, 12), each=3) dim(express)
有意な発現変化を起こしたものを拾ってくる。
参考にしたものでは、t検定を 0h vs (3h, 6h, 12h) で行い、すべての時間で有意に変化したものを有意と取っている。
ここから多重検定の話が絡んでくるが、面倒くさいので有意水準を0.0005にしている。
どれかひとつで有意に変化しているとかならANOVAでやったりする論文もある。
library(genefilter) alpha <- 0.0005 sig0 <- matrix(0, nr=nrow(express), nc=3) for(i in seq(3)){ label0 <- factor(rep(c(0, c(3, 6, 12)[i]), each=3)) sig0[, i] <- rowttests(express[, colnames(express) == 0 | colnames(express) == c(3, 6, 12)[i]], label0)$p.value } # すべての時間で有意なプローブをとってくる。 sig_all <- which(apply(sig0 < alpha, 1, all))
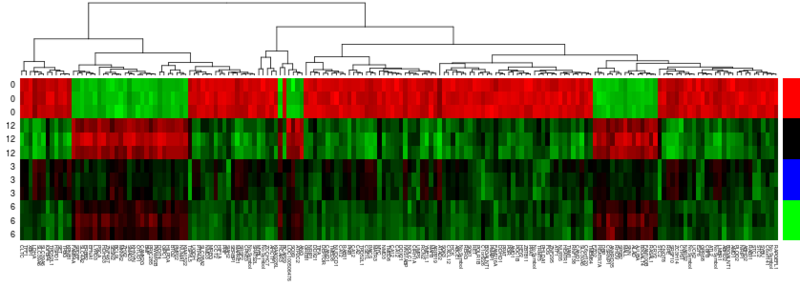
有意なものがとれたら、毎度おなじみのクラスタリングとヒートマップを描く。
順番としては前後するが、有意な遺伝子のレポートを作る工程を持ってきて、プローブIDを遺伝子と対応させておく。
library(gplots) library(annaffy) sig_probe <- rownames(express[sig_all, ]) report0 <- aafTableAnn(sig_probe, "hgu133plus2.db") saveText(report0, "report.txt") cols <- redgreen(100) csc <- rep(c("red", "blue", "green", "black"), each=3) express0 <- express[sig_all, ] rownames(express0) <- report0$Symbol rownames(express0)[rownames(express0) == "character(0)"] <- "No Symbol" heatmap.2(express0, col=cols, ColSideColors=csc, scale="row", trace="none", dendrogram="row", key=FALSE)
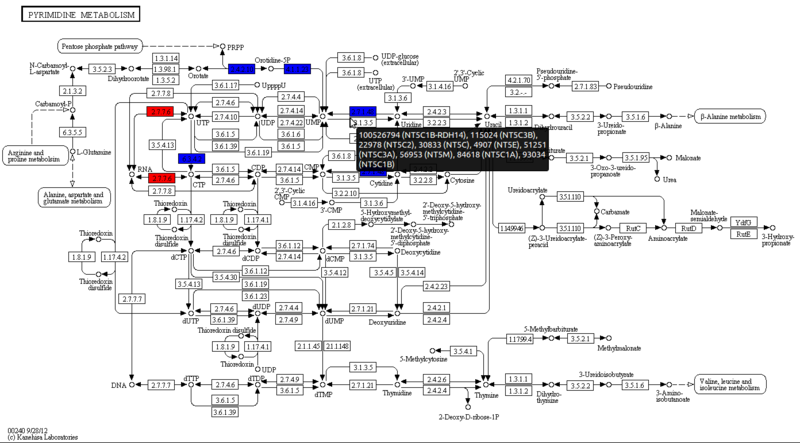
KEGGによるパスウェイ解析に続く。超幾何分布がうんたらかんたらはリンクを読む。
0hと3hを比較して、平均発現変化量が2倍(で1の差)があるかどうかでパスウェイ上に図示する。
# KEGG pathway analysis sig_probe <- rownames(express[sig_all, ]) sig_path <- na.omit(unique(unlist(mget(sig_probe, hgu133plus2PATH)))) res_path <- matrix(0, nr=length(sig_path), nc=4, dimnames=list(sig_path, c("expect", "observe", "total", "p.value"))) for(i in seq(sig_path)){ all_probe <- unlist(mget(sig_path[i], hgu133plus2PATH2PROBE)) res_path[i, "total"] <- length(all_probe) res_path[i, "observe"] <- length(intersect(sig_probe, all_probe)) res_path[i, "expect"] <- res_path[i, "total"]*length(sig_probe)/nrow(express) res_path[i, "p.value"] <- phyper(res_path[i, "observe"], res_path[i, "total"], nrow(express)-res_path[i, "total"], length(sig_probe), lower.tail=FALSE) } head(res_path[order(res_path[, "p.value"]), ]) obs_probe <- intersect(sig_probe, unlist(mget("00240", hgu133plus2PATH2PROBE))) gene_id <- unlist(mget(obs_probe, hgu133plus2ENTREZID)) M0 <- apply(apply(express[names(gene_id), 1:6], 1, tapply, rep(c(0, 3), each=3), mean), 2, diff) # 変化量 color <- c("red", "blue")[(M0 < 1) + 1] # 2倍以上の変化かそうでないかで色付け kegg_map <- data.frame(gene=gene_id, Color=color) write.table(kegg_map, "kegg_map.txt", quote=FALSE, append=TRUE, row.names=FALSE, col.names=FALSE)
kegg_map.txtができたら、KEGG Mapper – Search&Color Pathwayに飛んで、このファイルをアップロードもしくはコピペして、Homo sapiensを忘れずに指定してエンター。
すると、
hsa00240 Pyrimidine metabolism - Homo sapiens (human) (4)
hsa01100 Metabolic pathways - Homo sapiens (human) (4)
というのが出てくるので、クリックしたらパスウェイが取得できる。
オントロジー解析に続く。
GOデータベースには大きく3つの階層構造が存在するらしく、
- MF(molecular function):遺伝子産物の機能や個々の“能力”
- BP(biological process):1つ以上の機能により達成される細胞内イベント
- CC(cellular component):遺伝子産物の局在位置などの情報
となっているので、これらの引数を設定してとりあえずやってみる。
# GO analysis library(GO) library(GOstats) sig_go <- mget(sig_probe, hgu133plus2GO) entrezid <- na.omit(unique(unlist(mget(sig_probe, hgu133plus2ENTREZID)))) entrezuniverse <- na.omit(unique(unlist(mget(rownames(express), hgu133plus2ENTREZID)))) GOs <- c("MF", "CC", "BP") GOres <- vector("list", 3) names(GOres) <- GOs for(go in seq(GOs)){ para <- new("GOHyperGParams", geneIds=entrezid, universeGeneIds=entrezuniverse, annotation="hgu133plus2", ontology=GOs[go], pvalueCutoff=0.05, conditional=FALSE, testDirection="over") GOres[[go]] <- summary(hyperGTest(para)) }
$MF GOMFID Pvalue OddsRatio ExpCount Count Size 1 GO:0003700 0.0001180206 2.849669 7.9741658 20 926 2 GO:0001071 0.0001215259 2.842952 7.9913886 20 928 3 GO:0000988 0.0002247254 3.511531 4.0904198 13 475 4 GO:0043565 0.0008450881 2.893833 5.3132400 14 617 5 GO:0016829 0.0016954390 5.104143 1.2658773 6 147 6 GO:0016831 0.0019325838 13.598619 0.2497309 3 29 Term 1 sequence-specific DNA binding transcription factor activity 2 nucleic acid binding transcription factor activity 3 protein binding transcription factor activity 4 sequence-specific DNA binding 5 lyase activity 6 carboxy-lyase activity $CC GOCCID Pvalue OddsRatio ExpCount Count Size 1 GO:0031981 8.543649e-05 2.279652 17.49564 34 1990 2 GO:0070013 1.081434e-04 2.150656 21.57502 39 2454 3 GO:0043233 1.589787e-04 2.104979 21.96186 39 2498 4 GO:0005654 2.084776e-04 2.387940 12.37003 26 1407 5 GO:0044428 2.159357e-04 2.100578 20.68705 37 2353 6 GO:0031974 2.287651e-04 2.061893 22.33991 39 2541 Term 1 nuclear lumen 2 intracellular organelle lumen 3 organelle lumen 4 nucleoplasm 5 nuclear part 6 membrane-enclosed lumen $BP GOBPID Pvalue OddsRatio ExpCount Count Size 1 GO:0009628 1.574424e-06 3.792430 6.46562706 21 718 2 GO:0070482 7.767826e-06 5.917300 2.08917197 11 232 3 GO:0071453 1.060148e-05 13.831795 0.50428289 6 56 4 GO:0003188 1.397356e-05 112.775000 0.05403031 3 6 5 GO:0006915 2.440426e-05 2.644813 12.99428948 29 1443 6 GO:0001666 2.615509e-05 5.698346 1.95409620 10 217 Term 1 response to abiotic stimulus 2 response to oxygen levels 3 cellular response to oxygen levels 4 heart valve formation 5 apoptotic process 6 response to hypoxia