という質問を受けたので1万年と2000年ぶりくらいに遺伝統計やった。
雰囲気としては、こんな感じの値を求めたい。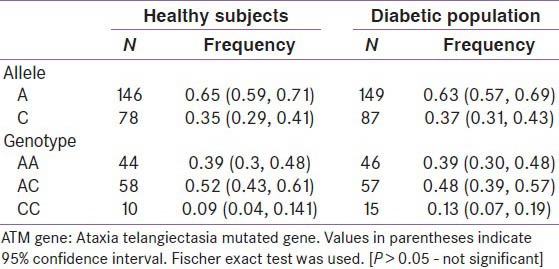
ここで、データとしては被験者たちのgenotypeを実験により決定(genotypingという)して、AA 44人、AC 58人、CC 10人という結果を得た。
まず、このgenotypeの頻度については、,
,
がそれぞれ人数を表していて、表記上
,
,
と対応させて、なおかつ
とする。
各genotype の頻度,
,
は
であるが、各genotype は多項分布からサンプリングされると仮定すると、多項分布の期待値は
, 分散は
である。
信頼区間は で求められるが、genotype が少ないとT分布くらいにしておいたら良さそうなので
x1 <- c(MM=44, MN=58, NN=10) x2 <- c(MM=46, MN=57, NN=15)
AlleleGenotypeFreqCalc(x1)$GenotypeFrequency
Frequency lowerCI upperCI MajorHomo 0.39285714 0.30142061 0.4842937 Hetero 0.51785714 0.42430584 0.6114084 MinorHomo 0.08928571 0.03589828 0.1426731
AlleleGenotypeFreqCalc(x2)$GenotypeFrequency
Frequency lowerCI upperCI MajorHomo 0.3898305 0.30092124 0.4787398 Hetero 0.4830508 0.39195381 0.5741479 MinorHomo 0.1271186 0.06639384 0.1878434
となりそれっぽくなった。
genotype は集団からgenotyping でデータを得られるが、実際に知りたいのはその元となったMとNが集団内にどれだけ存在しているかである。
遺伝学的には、MM、MN、NNの個体が交配することで、生物学的にはMとNを切り離し、確率的にはランダムで父方と母方のMもしくはNをもらうことで、次世代の個体のMMかMNかNNとなり、それが観測される。
MM、MN、NNの個体の数からMとNの数を推定するのは、HWEを利用すれば可能で、例えば
ここ(PDF)では、アレルの頻度(小文字で、なおかつこのページの上部のgenotype の頻度で使った
とは違うことに注意)は、genotype の頻度
(大文字)より
の分散は
(小文字と大文字に注意)
になる。
AlleleGenotypeFreqCalc(x1)$AlleleFrequency
Frequency lowerCI upperCI Major 0.6517857 0.5939582 0.7096132 Minor 0.3482143 0.2903868 0.4060418
AlleleGenotypeFreqCalc(x2)$AlleleFrequency
Frequency lowerCI upperCI Major 0.6313559 0.5709773 0.6917345 Minor 0.3686441 0.3082655 0.4290227
library(HardyWeinberg) alpha <- 0.05 AlleleGenotypeFreqCalc <- function(x=c(MM=44, MN=58, NN=10), alpha=0.05){ HW.test <- HWChisq(x, verbose=FALSE) # アレル頻度 AlleleFreq <- c(1-HW.test$p, HW.test$p) V2 <- (AlleleFreq + x[-2]/sum(x) - 2*AlleleFreq^2)/(2*sum(x)) AlleleFreqCI <- cbind(AlleleFreq, AlleleFreq-qnorm(1-alpha/2)*sqrt(V2), AlleleFreq+qnorm(1-alpha/2)*sqrt(V2) ) rownames(AlleleFreqCI) <- c("Major", "Minor") colnames(AlleleFreqCI) <- c("Frequency", "lowerCI", "upperCI") # HWEからの頻度 COV <- 2*V2[1]*(1+HW.test$f) GenotypeFreq <- c((1-HW.test$p)^2, 2*(1-HW.test$p)*HW.test$p, HW.test$p^2) GenotypeFreqCI <- cbind(GenotypeFreq, GenotypeFreq-qnorm(1-alpha/2)*sqrt(COV), GenotypeFreq+qnorm(1-alpha/2)*sqrt(COV) ) rownames(GenotypeFreqCI) <- c("MajorHomo", "Hetero", "MinorHomo") colnames(GenotypeFreqCI) <- c("Frequency", "lowerCI", "upperCI") # 多項分布からの頻度 MultinomP <- x/sum(x) CI <- qt(1-alpha/2, sum(x))*sqrt(MultinomP*(1-MultinomP)/sum(x)) MultinomFreq <- cbind(MultinomP, MultinomP-CI, MultinomP+CI) rownames(MultinomFreq) <- c("MajorHomo", "Hetero", "MinorHomo") colnames(MultinomFreq) <- c("Frequency", "lowerCI", "upperCI") return(list(input=x, AlleleFrequency=AlleleFreqCI, HWEFrequency=GenotypeFreqCI, GenotypeFrequency=MultinomFreq)) } x1 <- c(MM=44, MN=58, NN=10) x2 <- c(MM=46, MN=57, NN=15) AlleleGenotypeFreqCalc(x1) AlleleGenotypeFreqCalc(x2)