
細胞でもなんでもいいが、視野内の物体の動きを軌跡として取得したい。
最近流行りのディープラーニングでは下調べが足りないがいろいろあるっぽい。しかし、これらの手法を使うにしても自前で開発するにしても、正解データと比較して性能評価が必要である。これをGround truth という。
共同研究者からjava ベースで動く軽快なプログラムをもらったが、java のコンパイルがうまくいかないのでR で作ってみた。
と思ったらjava が動いたのでこれはお蔵入り。動画データはこちらからパクった。

R でこれをやろうと思ったら、画像を表示して、対象の細胞をクリックして挫傷を取得し、1枚画像を送って、最後までやる、を繰り返すことになる。
R では画像をポチポチして座標を取得するのに、locator 関数が使える。
これで細胞の座標を取得できるが、ひだりクリックで座標を取得し、みぎクリックは空打ちになるようなので、このクリック操作により条件分岐ができる。
追加したい機能としては、いままでにクリックした細胞はチェックしてわかりやすくすることと、失敗したらやり直すことである。
いままでにクリックした細胞は、履歴を取っておいて画像に重ね書きすることでできる。
(小さいがバツ印がついている)
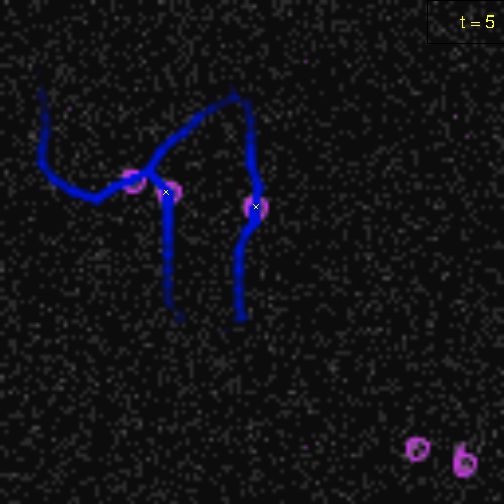
失敗したらやり直すのはちょっとめんどうだったので、画像をすべてのタイムフレームでクリックしたあとに気に入らなかったら全部捨てる仕様になっている。
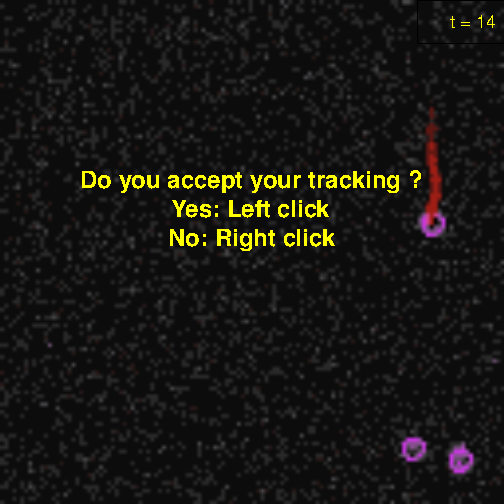
出力は細胞ごとにリストで、タイムフレームごとでのピクセル単位の(重心)座標が返ってくる。集中力が続く限り連続してやれる。

[[1]] [,1] [,2] [,3] [1,] 1 64.97317 6.472511 [2,] 2 63.64829 19.304606 [3,] 3 59.67362 25.343239 [4,] 4 64.02683 38.552749 [5,] 5 65.54098 53.083210 [6,] 6 61.18778 68.934621 [7,] 7 61.18778 81.011887 [8,] 8 53.04918 92.145617 [9,] 9 53.23845 101.203566 [10,] 10 53.80626 108.940565 [11,] 11 53.80626 118.375929 [12,] 12 NA NA [13,] 13 NA NA [14,] 14 NA NA [[2]] [,1] [,2] [,3] [1,] 1 65.35171 6.283804 [2,] 2 64.02683 18.738484 [3,] 3 59.10581 24.965825 [4,] 4 46.61401 34.401189 [5,] 5 42.82861 49.497771 [6,] 6 44.15350 61.386330 [7,] 7 43.01788 77.237741 [8,] 8 NA NA [9,] 9 NA NA [10,] 10 NA NA [11,] 11 NA NA [12,] 12 NA NA [13,] 13 NA NA [14,] 14 NA NA
この出力を再利用すれば、途中から作業を再開することができる。ただし、履歴が増えすぎると重い。
あとはバッチ処理にしてしまおうと思ったが、バッチ処理ではplot したときにプロットGUI が出てこないので諦めている。
Ground truth が必要といったが、この作業をひたすらやるのも諦めている。
### # # Rscript --slave --vanilla trackerR.R args0 args1 args2 # ### library(EBImage) library(png) library(jpeg) args <- commandArgs(TRUE) if(length(args) < 2){ cat( "command line args are args[0]: image sequence folder. format is currently .png args[1]: output filename. space separated txt. ID,t,x,y args[2]: optional. filepath of temporaly result of tracking. you can restart from this file.\n" ) do <- FALSE } else if (length(args) > 3){ cat( "args is less than 4.\n" ) do <- FALSE } else { do <- TRUE } input_dir <- args[1] out_name <- args[2] if(length(args) == 3){ tmp_res <- read.table(args[3], header=TRUE) final <- split(tmp_res[,-1], tmp_res$ID) } else { final <- list() } txt0 <- "Do you continue next tracking ?\nYes: Left click\nNo: Right click" txt1 <- "Do you accept your tracking ?\nYes: Left click\nNo: Right click" imgs <- list.files(input_dir, full.names=TRUE) ans <- TRUE accept <- FALSE if(do){ while(ans){ while(!accept){ res <- matrix(0, length(imgs), 3) for(i in seq(imgs)){ f <- readImage(imgs[i]) xleft <- 1 ybottom <- nrow(f) xright <- ncol(f) ytop <- 1 par(mar=rep(0, 4)) image(t(0), xlim=c(xleft, xright), ylim=c(ybottom, ytop), col="white") rasterImage(f, xleft, ybottom, xright, ytop) legend("topright", legend=paste0("t = ", i), text.col="yellow", cex=1.5) if(length(final) > 0){ for(j in seq(final)){ points(final[[j]][i, 2], final[[j]][i, 3], col="yellow", pch=4, cex=1, font=2) } } xy <- unlist(locator(1)) res[i, ] <- switch(is.numeric(xy)+1, c(i, NA, NA), c(i, xy)) } text((xleft+xright)/2, (ybottom+ytop)/2, txt1, col="yellow", pos=3, cex=2, font=2) accept <- is.list(locator(1)) } final <- c(final, list(res)) image(t(0), xlim=c(xleft, xright), ylim=c(ybottom, ytop), col="white") rasterImage(f, xleft, ybottom, xright, ytop) text((xleft+xright)/2, (ybottom+ytop)/2, txt0, col="yellow", pos=3, cex=2, font=2) ans <- is.list(locator(1)) accept <- FALSE } output <- cbind(rep(seq(final), each=length(imgs)), do.call(rbind, final)) colnames(output) <- c("ID", "t", "x", "y") write.table(output, out_name, quote=FALSE, row.names=FALSE) }