読んだ。
www.ncbi.nlm.nih.gov
巷を賑わせているCOVID-19だが、厚生労働省がダイヤモンド・プリンセス号のPCR陽性者数を逐一ネットに挙げていたので、この論文にもあるようにそこからデータを取ってきて、COVID-19の感染力を推定しようと思った。
Basic reproduction number - Wikipedia
ここで、感染症の感染力とは、いろいろゴニョゴニョやった結果、 と呼ばれる、感染者ひとりが何人に感染させるか、という推定値で感染力の強さがわかる。ここで、この論文での推定値は
(95% CI 2.06-2.52) ということになっている。
COVID-19は飛沫感染ということになっているので、だいたい1-3くらいの である。例えば、最強の空気感染の感染形態をもつ麻疹は、
=18 くらいあるようなので、めっちゃ強い(小並感
ちなみに、感染力 がわかると、集団のどれくらいの人が免疫をつければ感染を押さえ込めるかがモデル的にわかって、
となる。
なら勝手に感染が収束するし、
なら少しは集団免疫がないと感染が大爆発する。季節性インフルエンザは
くらいなので、60-70% くらいがワクチン接種をして集団免疫をつけると感染の大流行が防げるし、感染力の強い麻疹は、95% くらいが集団免疫をつけないと大流行する。
昔、こんなことをやった。
mikuhatsune.hatenadiary.com
さて、論文のとおりにやってみよう。論文ではまず、日本に停泊したダイヤモンド・プリンセス号のPCR陽性者数を、厚生労働省の発表から取ってきたようである。しかし、第8報(なぜか全角である)だけ引用されていて、停泊して隔離(?)され始めた2月3日から2月16日までのデータを利用したようだが、正直データは集まらないと思う。
横浜港に寄港したクルーズ船内で確認された新型コロナウイルス感染症について
新型コロナウイルス感染症の現在の状況と厚生労働省の対応について(令和2年2月6日版)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第3報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第4報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第5報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第6報)
新型コロナウイルス感染症の現在の状況と厚生労働省の対応について(令和2年2月12日版)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第8報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第9報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第10報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第11報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第12報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第13報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第14報)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(2月23日公表分)
横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(2月26日公表分)
実はwiki にまとまっていた。
クルーズ客船における2019年コロナウイルス感染症の流行状況 - Wikipedia
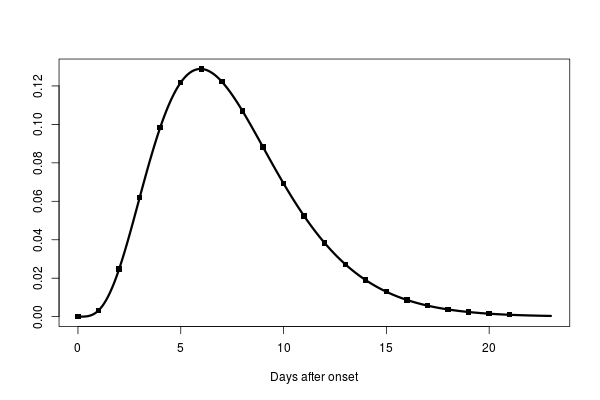
論文ではearlyR というパッケージのget_Rという関数で の推定を行なっている。これには感染から発症までの期間 serial interval というものが必要らしく、ガンマ分布で近似しているようなので、こちらの論文から平均 7.5、標準偏差 3.4 となるようにすると、
shape=4.865917、scale=1.541333 となる。
Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia. - PubMed - NCBI
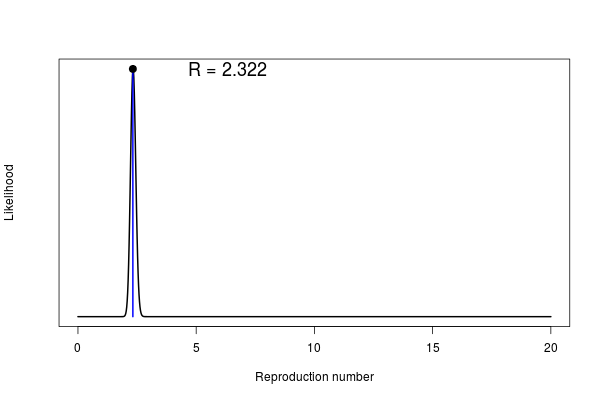
2月16日までのデータで論文の通り(? データがあっているのかわからないしコードも本当にあっているのかわからないのでわからない)に実行すると、=2.322 だった。
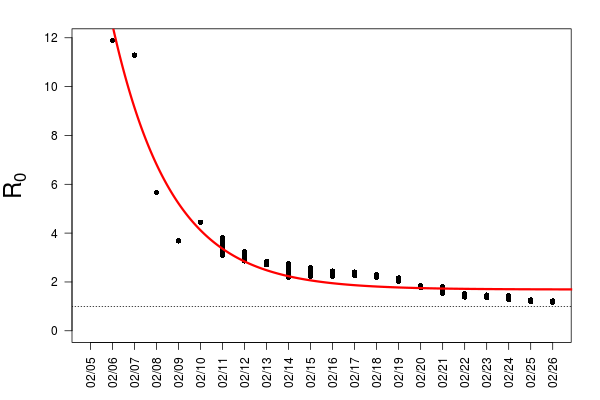
さて、ダイヤモンド・プリンセス号のデータは2月26日まであって、プロットをみる限りでは2月20日くらいから感染者数の急激な増加は収まっているようにみえるので、2月26日までのデータでやってみる。ここで、PCR陽性が判明した2月5日から、1日ずつ増加させてみながら を推定すると、感染拡大の超初期は莫大に感染が増えている(
が大きい)が、なんらかの値に収束していくようである。こういうように
を推定するのが果たして正しいのかどうかは知らない。
ここで、 の収束にはdrc パッケージの
EXD.3 関数を用い、漸近モデルとした。このとき、=1.62 くらいだった。
(飛んでいる日は前後の感染者総数および検査総数から適当に補完してシミュレーションしたので、若干のばらつきがある)
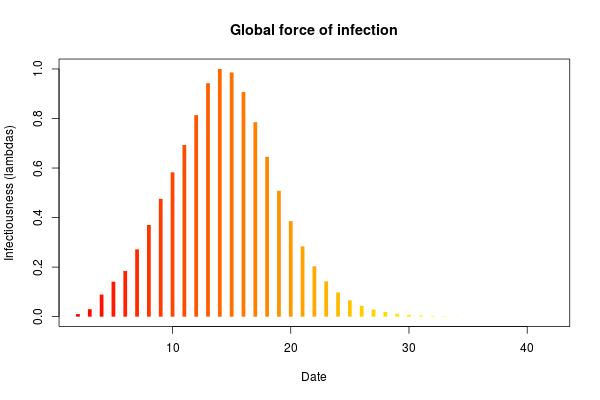
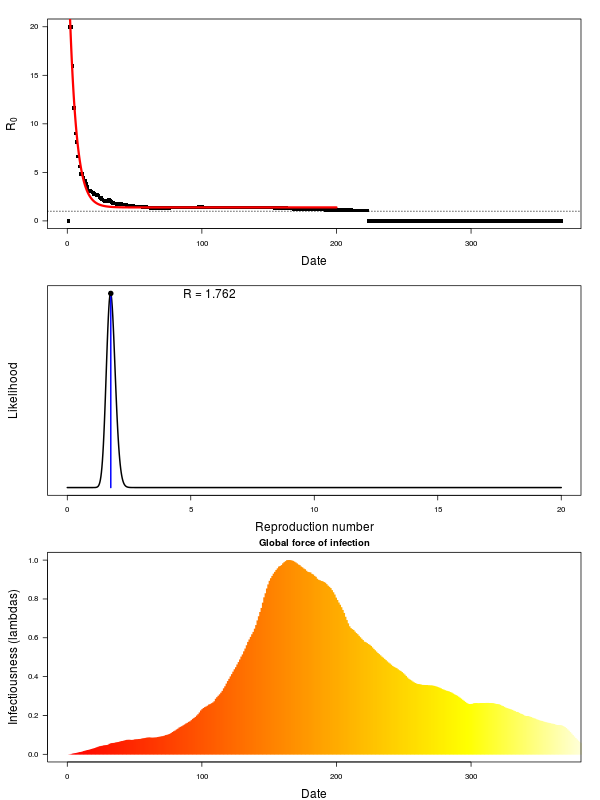
get_R の仕様をまだよく読み込んでないのだが、 というのが感染の勢い(?) を表している。ここでは観察から14日目(2月19日)にピークで、その後感染拡大の勢いが小さくなっている。
本当にこれであっているのかよくわからないので、サンプルでエボラをやってみた。2014年にエボラの大流行があって、outbreaks というパッケージにデータが入っている。エボラ自体は血液感染で、=2 くらいらしい。
これもCOVID-19 と同じようにやってみると、結局=1.5 くらいになった。いいのかよくわからない。
積んでるリスト
Time-varying transmission dynamics of Novel Coronavirus Pneumonia in China | bioRxiv
www.ncbi.nlm.nih.gov
https://web.stanford.edu/~jhj1/teachingdocs/Jones-on-R0.pdf
dat <- read.delim("corona_infection.txt", stringsAsFactors=FALSE) library(earlyR) library(incidence) library(drc) library(stringr) library(outbreaks) # 論文のデータを再現する xx <- seq(0, 23, length=1000) x <- 0:21 m0 <- 7.5 sd0 <- 3.4 # ガンマ分布のパラメータに戻す a <- (m0/sd0)^2 s <- (sd0^2/m0) plot(xx, dgamma(xx, shape=a, scale=s), type="l", xlab="Days after onset", ylab="", lwd=3) points(x, dgamma(x, shape=a, scale=s), pch=15) # 厚生労働省のデータは飛んでいる日があるので # 飛んでいる前後の日のデータから適当にサンプリングして補完する n <- dat$total_positive positive_N_simulator <- function(n){ r <- rle(is.na(n)) r0 <- tapply(n, rep(seq(r$lengths), r$lengths), c) r1 <- tapply(seq(n), rep(seq(r0), sapply(r0, length)), c) r2 <- sapply(sapply(r0, is.na), any) r3 <- sapply(r1[r2], length) r4 <- list(n[sapply(r1[r2], head, 1) - 1], n[sapply(r1[r2], tail, 1) + 1]) r5 <- sapply(mapply(function(z) sample(r4[[1]][z]:r4[[2]][z], size=r3[z], replace=FALSE), seq(r3)), sort) n1 <- replace(n, is.na(n), unlist(r5)) n2 <- mapply(rbinom, n=1, size=unlist(r5), prob=sample(na.omit(dat$total_positive/dat$total_n), size=1)) N <- replace(dat$test_positive, is.na(dat$test_positive), diff(n1)[which(is.na(dat$test_positive))-1]) return(N) } # wiki からとったデータ total <- 3711 wiki <- c(10, 20, 61, 64, 70, 135, 135, 174, 218, 218, 285, 355, 454, 542, 621, 634, 691, 705, 706, 706, 712) N <- c(wiki[1], diff(wiki)) # 飛んでいる日を適当に補完してシミュレーション iter <- 50 R0 <- matrix(NA, nrow(dat), iter) for(j in seq(iter)){ N <- positive_N_simulator(dat$total_positive) for(i in 2:length(N)){ inc <- incidence(rep(seq(N[1:i]), N[1:i])) res <- get_R(inc, si_mean=m0, si_sd=sd0, max_R=20) R0[i,j] <- res$R_ml } } # フィッテング Y <- c(R0) X <- c(row(R0)) fit <- drm(Y ~ X, fct=EXD.3()) cur <- predict(fit, as.data.frame(xx)) yl <- c(0, max(R0, na.rm=TRUE)) txt <- format(as.Date(dat$date), "%m/%d") rownames(R0) <- txt par(mar=c(4, 5, 2, 2)) matplot(R0, pch=16, xaxt="n", ylim=yl, las=2, xlab="", ylab=expression(R[0]), col=1, cex.lab=2) axis(1, at=seq(nrow(R0)), labels=txt, las=2) abline(h=1, lty=3) lines(xx, cur, lwd=3, col="red") # エボラでやってみる onset <- outbreaks::ebola_sim$linelist$date_of_onset N <- table(onset) R0 <- rep(0, length(N)) res <- vector("list", length(N)) for(i in 2:length(R0)){ inc <- incidence(rep(seq(N[1:i]), N[1:i])) res[[i]] <- get_R(inc, disease="ebola", max_R=20) R0[i] <- res[[i]]$R_ml } xx <- seq(0, 200, length=1000) Y <- R0[R0 > 0] X <- seq(R0)[R0 > 0] fit <- drm(Y ~ X, fct=EXD.3()) cur <- predict(fit, as.data.frame(xx)) xl <- c(0, length(N)) par(mfrow=c(3, 1), mar=c(4, 5, 2, 2), cex.lab=1.5, las=1) plot(R0, xlab="Date", ylab=expression(R[0]), xlim=xl, pch=15) lines(xx, cur, lwd=3, col="red") abline(h=1, lty=3) plot(res[[40]]) # 適当に40日目 plot(tail(res, 1)[[1]], "lambda", xlim=xl)
date test_positive test_n asymptomatic total_positive total_n total_asymptomatic title url 2020-02-05 10 31 10 31 横浜港に寄港したクルーズ船内で確認された新型コロナウイルス感染症について https://www.mhlw.go.jp/stf/newpage_09276.html 2020-02-06 10 71 20 102 新型コロナウイルス感染症の現在の状況と厚生労働省の対応について(令和2年2月6日版) https://www.mhlw.go.jp/stf/newpage_09360.html 2020-02-07 41 171 61 273 横浜港に寄港したクルーズ船内で確認された新型コロナウイルス感染症について https://www.mhlw.go.jp/stf/newpage_09340.html 2020-02-08 3 6 64 279 横浜港に寄港したクルーズ船内で確認された新型コロナウイルス感染症について(第4報) https://www.mhlw.go.jp/stf/newpage_09398.html 2020-02-09 6 57 70 336 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第5報) https://www.mhlw.go.jp/stf/newpage_09405.html 2020-02-10 65 103 135 439 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第6報) https://www.mhlw.go.jp/stf/newpage_09419.html 2020-02-11 2020-02-12 39 67 174 492 新型コロナウイルス感染症の現在の状況と厚生労働省の対応について(令和2年2月12日版) https://www.mhlw.go.jp/stf/newpage_09450.html 2020-02-13 44 221 218 713 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第8報) https://www.mhlw.go.jp/stf/newpage_09425.html 2020-02-14 2020-02-15 67 217 38 285 930 73 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第9報) https://www.mhlw.go.jp/stf/newpage_09542.html 2020-02-16 70 289 38 355 1219 111 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第10報) https://www.mhlw.go.jp/stf/newpage_09547.html 2020-02-17 99 504 70 454 1723 189 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第11報) https://www.mhlw.go.jp/stf/newpage_09568.html 2020-02-18 88 681 65 542 2404 254 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第12報) https://www.mhlw.go.jp/stf/newpage_09606.html 2020-02-19 79 607 68 621 3011 322 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第13報) https://www.mhlw.go.jp/stf/newpage_09640.html 2020-02-20 13 52 6 634 3063 328 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(第14報) https://www.mhlw.go.jp/stf/newpage_09668.html 2020-02-21 2020-02-22 2020-02-23 57 831 52 691 3894 380 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(2月23日公表分) https://www.mhlw.go.jp/stf/newpage_09708.html 2020-02-24 2020-02-25 2020-02-26 14 167 12 705 4061 392 横浜港で検疫中のクルーズ船内で確認された新型コロナウイルス感染症について(2月26日公表分) https://www.mhlw.go.jp/stf/newpage_09783.html