遺伝子座による形質の定量度の違いをquantitative trait loci (QTL)というらしい。
普通、ある遺伝子座がA→Gだと、収縮期血圧が10mmHg高い傾向にある、みたいな、平均値の変動を定量化することが多いが、分散の変動、つまり、普通アレルだと平均±5mmHgのところが、このアレルだと±10mmHgでばらつく、みたいな影響のことを variance QTL というらしい。
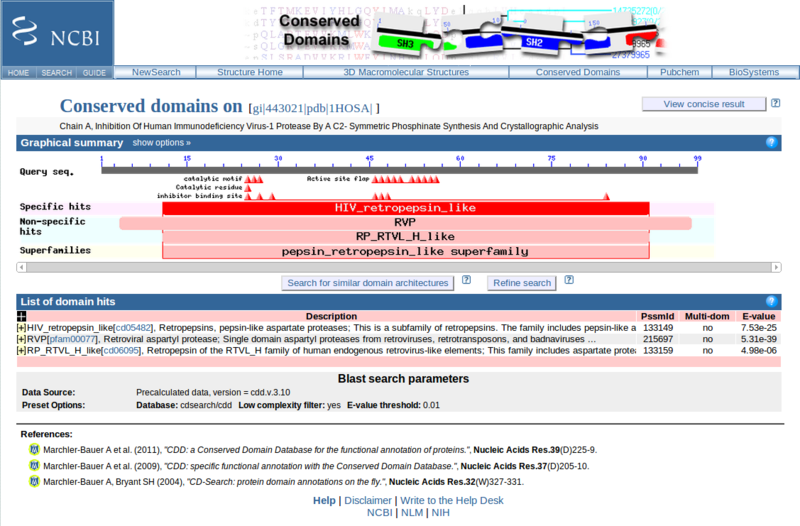
Shen X., et al. PloS Genet. 2012.では、Arabidopsis thaliana(シロイヌナズナ)のデータベースから、モリブデン輸送体MOT1のgenotypeとモリブデン濃度のばらつきを示している。
論文Figure 3より引用

vQTLについての手法はRönnegård L. BMC Genet 2012.にいくつかまとまっている。
論文Figure 1より引用。

Rではqtl, QTLRelパッケージが検索でひっかかるが、中身をじっくり読むヒマがないので、プロット的なことをしてくれるものをやってみたら、マンハッタンプロット的なものが出てきたけどよくわからないので保留。
data(miscEx) # impute missing genotypes gdat.imp <- genoImpute(gdatF8, gmap=gmapF8, step=Inf, gr=8, na.str=NA) # estimate variance componentsqqPlot 27 o <- estVC(y=pdatF8$bwt, x=pdatF8$sex, v=list(AA=gmF8$AA,DD=gmF8$DD, HH=NULL, AD=NULL, MH=NULL, EE=diag(length(pdatF8$bwt)))) # genome scan llk <- scanOne(y=pdatF8$bwt, x=pdatF8$sex, vc=o, gdat=gdat.imp) # plotting plot(llk, gmap=gmapF8) # gmap is needed # plotting in another way idx <- match(colnames(gdat.imp), gmapF8$snp) tmp <- data.frame(chr=gmapF8$chr[idx],dist=gmapF8$dist[idx],y=llk$p) plotit(tmp, main="Mapping Plot", xlab="Chromosome", ylab="LRT", col=as.integer(tmp$ch)%%2+2,type="p")