検定とp値がいつまで経ってもわからないので久々にシミュレーションした。
対照群 と治療群
があって、なんらかの指標となる値に変化があるかどうか調べたい。
比較するのは、母集団(真の分布)である(ここでは真の分布は正規分布であると自分は分かっているので、 は自分で決められるしシミュレーションでは正確に比較検討できる)の平均
である。
ここではシミュレーションなので は分かっているが、実際のデータ解析においては母集団(真の分布)は分布の形もわからないしもちろん真のパラメータの値もわからないのが、実験して標本(サンプル)は分かる。この標本のデータを使って
帰無仮説 と対立仮説
を考えるのが一般的な帰無仮説検定とp値の扱いである。
が真と仮定してp値を計算したらp<0.05 なのでこれは棄却(本当は有意水準
)...というのがよくある考え方である。
単純に総当りとして が真か偽で、それぞれp>0.05 で棄却しないもしくはp<0.05 で棄却するパターンの2x2 通りをシミュレーションした。
B:帰無仮説 が真(本当に差がない)のにも関わらず、p<0.05 となり棄却する、というのは、p値の定義からは確率論的にありうるので、統計学的には正しい。しかし実践医学的には正しくはない。
C:帰無仮説 が偽(差があることが神視点では分かっている)なのに、p<0.05 にならずに帰無仮説が棄却されない、というのも、統計学的にはありえる正しい結果である。これは検出力による。だがこれも実践医学的には正しくない、というか好ましくない。
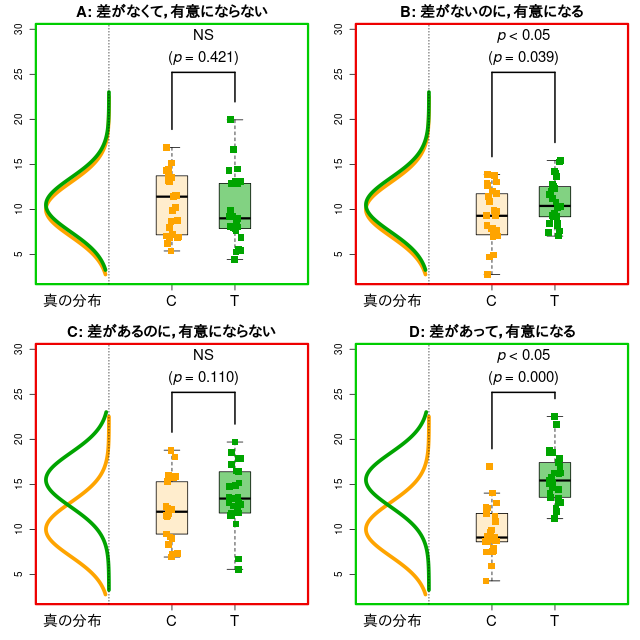
n <- 25 # サンプルサイズ g <- rep(1:2, each=n) alpha <- 0.05 dist_q <- 0.01 # プロット用の真の分布の上下限の分位点% # 真の分布を正規分布から ms <- c(10, 15) sds <- 3 # シミュレーションデータをつくる set.seed(1234) flag <- TRUE while(flag){ y <- list( mapply(rnorm, n, ms[c(1, 1)], sds, SIMPLIFY=FALSE), mapply(rnorm, n, ms[c(1, 1)], sds, SIMPLIFY=FALSE), mapply(rnorm, n, ms[c(1, 2)], sds, SIMPLIFY=FALSE), mapply(rnorm, n, ms[c(1, 2)], sds, SIMPLIFY=FALSE) ) pv <- mapply(function(z) t.test(z ~ g)$p.value, lapply(y, unlist)) flag <- !(pv[1] > alpha & pv[2] < alpha & pv[3] > alpha & pv[4] < alpha) } for(i in seq(y)) names(y[[i]]) <- c("C", "T") # 一気に見つけるにはよいが # 4パターン同時に見つけるには対立仮説が真のときに帰無仮説が棄却されないパターンがなかなか見つからなくなるので個別にシミュレーションデータを作る flag <- TRUE while(flag){ tmp0 <- mapply(rnorm, n, ms[c(1, 1)], sds, SIMPLIFY=FALSE) p0 <- t.test(unlist(tmp0) ~ g)$p.value flag <- !(p0 > alpha) } flag <- TRUE while(flag){ tmp1 <- mapply(rnorm, n, ms[c(1, 1)], sds, SIMPLIFY=FALSE) p1 <- t.test(unlist(tmp1) ~ g)$p.value flag <- !(p1 < alpha) } flag <- TRUE while(flag){ tmp2 <- mapply(rnorm, n, ms[c(1, 2)], sds, SIMPLIFY=FALSE) p2 <- t.test(unlist(tmp2) ~ g)$p.value flag <- !(p2 > alpha) } flag <- TRUE while(flag){ tmp3 <- mapply(rnorm, n, ms[c(1, 2)], sds, SIMPLIFY=FALSE) p3 <- t.test(unlist(tmp3) ~ g)$p.value flag <- !(p3 < alpha) } y <- list(tmp0, tmp1, tmp2, tmp3) for(i in seq(y)) names(y[[i]]) <- c("C", "T") pv <- c(p0, p1, p2, p3) cols <- c("orange", "green2") colsa <- c(rgb(255, 165, 0, 50, maxColorValue=256), rgb(0, 165, 0, 125, maxColorValue=256)) cols <- gsub(".{2}$", "", colsa) dx <- 0.3 xl <- c(-1, 3) yl <- range(unlist(y)) yl <- range(mapply(function(z) mapply(qnorm, z, ms, sds), c(dist_q, 1-dist_q)), unlist(y)) x <- seq(yl[1], yl[2], length=3000) # 真の分布のプロット用 Y <- list( mapply(dnorm, list(x), ms[c(1, 1)], sds, SIMPLIFY=FALSE), mapply(dnorm, list(x), ms[c(1, 1)], sds, SIMPLIFY=FALSE), mapply(dnorm, list(x), ms[c(1, 2)], sds, SIMPLIFY=FALSE), mapply(dnorm, list(x), ms[c(1, 2)], sds, SIMPLIFY=FALSE) ) Y <- rapply(Y, function(z) -z*1/max(unlist(Y)), how="replace") dY <- -10 # Y <- rapply(Y, function(z) z*dY, how="replace") dx <- 0.5 # 帰無仮説が真のときにプロットを少しずらす marginY <- 7 marginy <- 2 text <- c("差がなくて,有意にならない", "差がないのに,有意になる", "差があるのに,有意にならない", "差があって,有意になる") text <- paste(LETTERS[seq(text)], text, sep=": ") bcols <- c("green3", "red2")[c(1, 2, 2, 1)] # png("significant.png", 640, 640) par(mfrow=c(2, 2), mar=c(3, 3, 2, 1)) for(j in seq(y)){ plot(0, type="n", xlim=xl, ylim=yl+c(0, marginY), frame=FALSE, xaxt="n", yaxt="n", xlab="", ylab="", main=text[j], cex.main=1.5) boxplot(y[[j]], at=1:2, add=TRUE, outline=FALSE, boxwex=0.5, col=colsa, frame=FALSE, xaxt="n", yaxt="n") axis(1, at=1:2, labels=names(y[[j]]), lwd=0, lwd.ticks=1, cex.axis=1.5) axis(1, at=(par()$usr[1]+0)/2, labels="真の分布", lwd.ticks=0, cex.axis=1.5) axis(2, lwd=0, lwd.ticks=1) box(col=bcols[j], lwd=3) stripchart(y[[j]], method="jitter", vertical=TRUE, add=TRUE, col=cols, pch=15, cex=1.5) for(i in 1:2) lines(Y[[j]][[i]], x+dx*(i-1), col=cols[i], lwd=5) abline(v=0, lty=3) yy <- sapply(y[[j]], max) # y2 <- (par()$usr[4] + max(yy))/2 # y2 <- (par()$usr[4] + max(unlist(y)))/2 y2 <- sum(c(par()$usr[4], max(unlist(y))) * c(1, 2)/3) for(i in 1:2) segments(i, y0=yy[i]+marginy, y1=y2, lwd=2) segments(x0=1, x1=2, y0=y2, lwd=2) if(pv[j] > alpha){ txt <- substitute(atop("NS", "("*italic(p)~"="~x*")"), list(x=sprintf("%.3f", pv[j]))) } else { txt <- substitute(atop(italic(p)~"<"~y, "("*italic(p)~"="~x*")"), list(x=sprintf("%.3f", pv[j]), y=alpha)) } text(1.5, y2, txt, pos=3, cex=1.5) } # dev.off()