Shinyというhtml 上でいろいろパラメータをいじったものを即座に計算して描出するインタラクティブでなんかすごいアプリがあったのだが、rmarkdown と同様に手をつけることなく放置していた。
せっかちな人はこちら → https://yfujii08.shinyapps.io/pkpdsimulator/
一番出来のいいエクセルは → https://home.hiroshima-u.ac.jp/r-nacamura/
話は変わるのだが、手術の最中に鎮痛(痛み止め)としてフェンタニルという麻薬を使う。この薬は適当に投与していてもなんとかなるが、本当にいい感じに使いたかったら、痛みがとれる濃度に収めつつ、かつ、副作用として呼吸抑制(息をしてくれない)を起こさない濃度にしなければならない。
そして、手術の最中は適当に投与していてもいい(手術中は人工呼吸器という機械で息をしてくれているのは保証されているから)が、手術が終わったあとも、上記のいい感じの濃度に保って鎮痛を継続して図りたいのである。
ここで、濃度のシミュレーションアプリとして、FentaSim(無料)、iTIVA anesthesia(無料といいながらアプリ内課金でぜんぜん無料で使えない)、AnestAssist PK/PD(潔く有料)というものがいくつか出ているのだが、これらのアプリがやってくれているのは、結局、
・濃度シミュレーション
・UI として可視化
というわけである。濃度シミュレーションはAnesthesiology. 1990 Dec;73(6):1091-102.あたりから必要な微分方程式とパラメータを持ってきている。要は3コンパートメントモデルと効果部位を想定して、コンパートメント から
から への移動を
への移動を と表記する。ここで効果部位は
と表記する。ここで効果部位は , メイン(というか静脈注射されて薬が一気に流入してくるコンパートメントのことで、たいてい1番目)コンパートメントから他のコンパートメント以外への排泄を0 として、使われるパラメータは
, メイン(というか静脈注射されて薬が一気に流入してくるコンパートメントのことで、たいてい1番目)コンパートメントから他のコンパートメント以外への排泄を0 として、使われるパラメータは ,
,  ,
,  ,
,  ,
,  ,
,  となる。
となる。
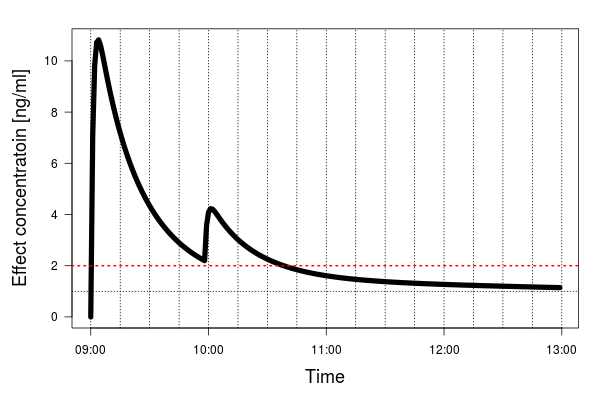
というわけでフェンタニルのパラメータを使って開始時と1時間のときに適当に投与した結果がこちら。
weight <- 50
height <- 160
gender <- "M"
LBM <- ifelse(gender == "M", 1.1*weight-128*(weight/height)^2,1.07*weight-148*(weight/height)^2)
timestep <- 120
k_1N <- c(0.0827, 0.471, 0.225)
k_N1 <- c(0.102, 0.006)
k_e0 <- 0.114
k <- c(k_1N, k_N1, k_e0)/(60/timestep)
names(k) <- c(sprintf("k1%d", c(0, 2, 3)), sprintf("k%d1", 2:3), "ke0")
k1 <- k["k10"] + k["k12"] + k["k13"]
V1 <- 0.105*weight
CLs <- k[c("k10", "k12", "k13")]*(60/timestep)*V1
V2 <- CLs[2]/(k["k21"]*(60/timestep))
V3 <- CLs[3]/(k["k31"]*(60/timestep))
X[1, "Bolus"] <- 123000*(60/timestep)
X[60, "Bolus"] <- 100000*(60/timestep)
X <- rbind(0, X)
for(i in 1:(nrow(X)-1)){
X$dA1_1[i+1] <- k["k21"]*X$A2[i]+k["k31"]*X$A3[i]-k1*X$A1[i]+X$Cont[i+1]/(60/timestep)
X$dA2_1[i+1] <- k["k12"]*X$A1[i] - k["k21"]*X$A2[i]
X$dA3_1[i+1] <- k["k13"]*X$A1[i] - k["k31"]*X$A3[i]
X$dAe_1[i+1] <- k["ke0"]*(X$A1[i] - X$Ae[i])
X$dA1_2[i+1] <- k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)-k1*(X$A1[i]+X$dA1_1[i+1]/2)+X$Cont[i+1]/(60/timestep)
X$dA2_2[i+1] <- k["k12"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)
X$dA3_2[i+1] <- k["k13"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)
X$dAe_2[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_1[i+1]/2 - (X$Ae[i]+X$dAe_1[i+1]/2))
X$dA1_3[i+1] <- k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)-k1*(X$A1[i]+X$dA1_2[i+1]/2)+X$Cont[i+1]/(60/timestep)
X$dA2_3[i+1] <- k["k12"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)
X$dA3_3[i+1] <- k["k13"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)
X$dAe_3[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_2[i+1]/2 - (X$Ae[i]+X$dAe_2[i+1]/2))
X$dA1_4[i+1] <- k["k21"]*(X$A2[i]+X$dA2_3[i+1])+k["k31"]*(X$A3[i]+X$dA3_3[i+1])-k1*(X$A1[i]+X$dA1_3[i+1])+X$Cont[i+1]/(60/timestep)
X$dA2_4[i+1] <- k["k12"]*(X$A1[i]+X$dA1_3[i+1]) - k["k21"]*(X$A2[i]+X$dA2_3[i+1])
X$dA3_4[i+1] <- k["k13"]*(X$A1[i]+X$dA1_3[i+1]) - k["k31"]*(X$A3[i]+X$dA3_3[i+1])
X$dAe_4[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_3[i+1] - (X$Ae[i]+X$dAe_3[i+1]))
X$A1[i+1] <- X$A1[i]+(X$dA1_1[i+1]+2*X$dA1_2[i+1]+2*X$dA1_3[i+1]+X$dA1_4[i+1])/6+X$Bolus[i+1]/(60/timestep)
X$A2[i+1] <- X$A2[i]+(X$dA2_1[i+1]+2*X$dA2_2[i+1]+2*X$dA2_3[i+1]+X$dA2_4[i+1])/6
X$A3[i+1] <- X$A3[i]+(X$dA3_1[i+1]+2*X$dA3_2[i+1]+2*X$dA3_3[i+1]+X$dA3_4[i+1])/6
X$Ae[i+1] <- X$Ae[i]+(X$dAe_1[i+1]+2*X$dAe_2[i+1]+2*X$dAe_3[i+1]+X$dAe_4[i+1])/6
X$Cp[i+1] <- X$A1[i+1]/V1/1000
X$Ce[i+1] <- X$Ae[i+1]/V1/1000
X$C1[i+1] <- X$A1[i+1]/V1/1000
X$C2[i+1] <- X$A2[i+1]/V2/1000
X$C3[i+1] <- X$A3[i+1]/V3/1000
}
X <- X[-1,]
t0 <- Sys.time()
tx <- t0 + (seq(nrow(X))-1)*60
yl <- c(0, max(X$Ce))
par(mar=c(5, 5, 1, 1), las=1)
plot(tx, X$Ce, type="l", ylim=yl)
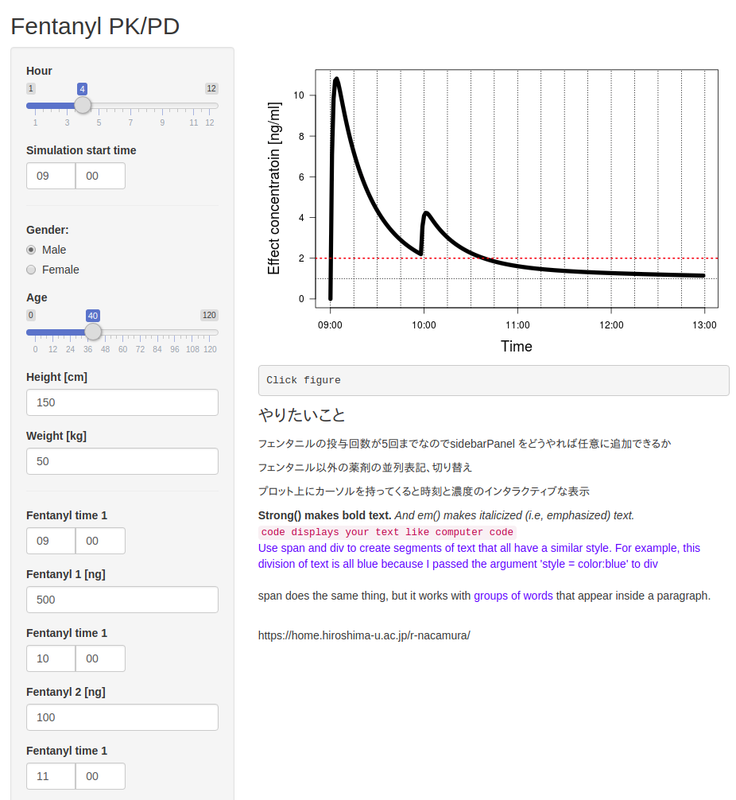
さて、自分のPCが手元にあるときは自由にパラメータを設定して任意にシミュレーションできるが、PCがないときやRを使わない他の人に使ってもらいたいときに困る。このようなときにアプリ化しておくと便利である。これはShiny でできる。
Shiny のサンプルはRstudio の新規作成 > shiny にすれば、ヒストグラムを描いてbin をインタラクティブに操作できるものが作成される。出力はoutput$hoge に入っていて、サンプルではoutput$distPlot でヒストグラムが翔ようになっている。ここをいじる。
output にはinput$bins から必要な変数が取得できるので、ここをいじる。生値を入力するのか、スライドバーで設定するのか、選択肢を作るのかはhogehogeInput 関数で設定できる。
アプリを作るにはui.R とapp.R を作る必要があるようだが、app.R 内にui とserver をごちゃごちゃ書きこめばなんとかなるっぽい。アプリを公開するには手っ取り早いのはshinyserver を使う。rmd をRpubs に公開するのと同じように、Rstudio から run app したあとに出力されるhtml の右上に公開、というボタンがあるのでそれを押すと、「アカウントある?」と聞かれるのでとりあえずアカウントを作る。アカウントを作成したあとは、アプリ公開のためのキーを取得して紐づけする必要があるので、SECRET を直して他人に見られないようにRにコピペする。これはツイッターAPI などを使う人はやったことがあるはず。
そのあと、Rstudio から公開しようとするとなぜかできなかった。なのでRコンソールから
rsconnect::deployApp('path/to/your/app')
とするとなぜかうまくいった。このとき、app はapp.R ではなく、app.R があるディレクトリを参照する。
library(shiny)
library(shinyTime)
ui <- fluidPage(
titlePanel("Fentanyl PK/PD"),
sidebarLayout(
sidebarPanel(
sliderInput("hour", "Hour", min = 1, max = 12, value = 4, step=1),
timeInput("starttime", label = "Simulation start time", value = strptime("09:00:00", "%T"), seconds=FALSE),
hr(),
radioButtons("gender", "Gender:", c("Male" = "M", "Female" = "F")),
sliderInput("age", label="Age", value = 40, min = 0, max = 120, step=1),
numericInput("height", label = "Height [cm]", min=1, max=250, value = 150),
numericInput("weight", label = "Weight [kg]", min=1, max=150, value = 50),
hr(),
timeInput("ftime1", label = "Fentanyl time 1", value = strptime("09:00:00", "%T"), seconds=FALSE),
numericInput("iv1", label = "Fentanyl 1 [ng]", min=0, max=1000, value = 50),
timeInput("ftime2", label = "Fentanyl time 2", value = strptime("10:00:00", "%T"), seconds=FALSE),
numericInput("iv2", label = "Fentanyl 2 [ng]", min=0, max=1000, value = 0),
timeInput("ftime3", label = "Fentanyl time 3", value = strptime("11:00:00", "%T"), seconds=FALSE),
numericInput("iv3", label = "Fentanyl 3 [ng]", min=0, max=1000, value = 0),
timeInput("ftime4", label = "Fentanyl time 4", value = strptime("12:00:00", "%T"), seconds=FALSE),
numericInput("iv4", label = "Fentanyl 4 [ng]", min=0, max=1000, value = 0),
timeInput("ftime5", label = "Fentanyl time 5", value = strptime("13:00:00", "%T"), seconds=FALSE),
numericInput("iv5", label = "Fentanyl 5 [ng]", min=0, max=1000, value = 0),
hr(),
sliderInput("ylim", label="ylim [ng/ml]", value = 0, min = 0, max = 5, step=0.5),
sliderInput("Cont_conc", label="ivPCA [ng/hr]", value = 0, min = 0, max = 50, step=5),
timeInput("ivPCAstart", label = "ivPCA start time", value = strptime("12:00:00", "%T"), seconds=FALSE),
hr(),
fluidRow(column(3, verbatimTextOutput("value")))
),
mainPanel(
plotOutput("distPlot", click="plot_click"),
verbatimTextOutput("info"),
p("やりたいこと", style = "font-family: 'times'; font-size: 16pt"),
p("フェンタニルの投与回数が5回までなのでsidebarPanel をどうやれば任意に追加できるか"),
p("フェンタニル以外の薬剤の並列表記、切り替え"),
p("プロット上にカーソルを持ってくると時刻と濃度のインタラクティブな表示"),
strong("Strong() makes bold text."),
em("And em() makes italicized (i.e, emphasized) text."),
br(),
code("code displays your text like computer code"),
div("Use span and div to create segments of text that all have a similar style. For example, this division of text is all blue because I passed the argument 'style = color:blue' to div", style = "color:blue"),
br(),
p("span does the same thing, but it works with",
span("groups of words", style = "color:blue"),
"that appear inside a paragraph."),
br(),
p("https://home.hiroshima-u.ac.jp/r-nacamura/")
)
)
)
server <- function(input, output) {
test <- 1
output$distPlot <- renderPlot({
weight <- input$weight
comp <- c(1:3, "e")
cname <- c("Bolus", "Cont", sprintf("A%s", comp), mapply(function(z) sprintf("dA%s_%d", comp, z), 1:4), "Cp", sprintf("C%s", comp))
X <- matrix(0, input$hour*60, length(cname), dimnames=list(NULL, cname))
X <- as.data.frame(X)
timestep <- 60
k_1N <- c(0.0827, 0.471, 0.225)
k_N1 <- c(0.102, 0.006)
k_e0 <- 0.114
k <- c(k_1N, k_N1, k_e0)/(60/timestep)
names(k) <- c(sprintf("k1%d", c(0, 2, 3)), sprintf("k%d1", 2:3), "ke0")
k1 <- k["k10"] + k["k12"] + k["k13"]
V1 <- 0.105*weight
CLs <- k[c("k10", "k12", "k13")]*(60/timestep)*V1
V2 <- CLs[2]/(k["k21"]*(60/timestep))
V3 <- CLs[3]/(k["k31"]*(60/timestep))
t0 <- input$starttime - 61*0
tx <- t0 + (seq(nrow(X))-1)*60
X[tx > input$ivPCAstart, "Cont"] <- input$Cont_conc/60*1000
X <- na.omit(X)
X <- rbind(0, X)
X[tail(which(tx < input$ftime1+61), 1), "Bolus"] <- input$iv1*1000*(60/timestep)
X[tail(which(tx < input$ftime2), 1), "Bolus"] <- input$iv2*1000*(60/timestep)
X[tail(which(tx < input$ftime3), 1), "Bolus"] <- input$iv3*1000*(60/timestep)
X[tail(which(tx < input$ftime4), 1), "Bolus"] <- input$iv4*1000*(60/timestep)
X[tail(which(tx < input$ftime5), 1), "Bolus"] <- input$iv5*1000*(60/timestep)
for(i in 1:(nrow(X)-1)){
X$dA1_1[i+1] <- k["k21"]*X$A2[i]+k["k31"]*X$A3[i]-k1*X$A1[i]+X$Cont[i+1]/(60/timestep)
X$dA2_1[i+1] <- k["k12"]*X$A1[i] - k["k21"]*X$A2[i]
X$dA3_1[i+1] <- k["k13"]*X$A1[i] - k["k31"]*X$A3[i]
X$dAe_1[i+1] <- k["ke0"]*(X$A1[i] - X$Ae[i])
X$dA1_2[i+1] <- k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)-k1*(X$A1[i]+X$dA1_1[i+1]/2)+X$Cont[i+1]/(60/timestep)
X$dA2_2[i+1] <- k["k12"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_1[i+1]/2)
X$dA3_2[i+1] <- k["k13"]*(X$A1[i]+X$dA1_1[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_1[i+1]/2)
X$dAe_2[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_1[i+1]/2 - (X$Ae[i]+X$dAe_1[i+1]/2))
X$dA1_3[i+1] <- k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)+k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)-k1*(X$A1[i]+X$dA1_2[i+1]/2)+X$Cont[i+1]/(60/timestep)
X$dA2_3[i+1] <- k["k12"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k21"]*(X$A2[i]+X$dA2_2[i+1]/2)
X$dA3_3[i+1] <- k["k13"]*(X$A1[i]+X$dA1_2[i+1]/2) - k["k31"]*(X$A3[i]+X$dA3_2[i+1]/2)
X$dAe_3[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_2[i+1]/2 - (X$Ae[i]+X$dAe_2[i+1]/2))
X$dA1_4[i+1] <- k["k21"]*(X$A2[i]+X$dA2_3[i+1])+k["k31"]*(X$A3[i]+X$dA3_3[i+1])-k1*(X$A1[i]+X$dA1_3[i+1])+X$Cont[i+1]/(60/timestep)
X$dA2_4[i+1] <- k["k12"]*(X$A1[i]+X$dA1_3[i+1]) - k["k21"]*(X$A2[i]+X$dA2_3[i+1])
X$dA3_4[i+1] <- k["k13"]*(X$A1[i]+X$dA1_3[i+1]) - k["k31"]*(X$A3[i]+X$dA3_3[i+1])
X$dAe_4[i+1] <- k["ke0"]*(X$A1[i]+X$dA1_3[i+1] - (X$Ae[i]+X$dAe_3[i+1]))
X$A1[i+1] <- X$A1[i]+(X$dA1_1[i+1]+2*X$dA1_2[i+1]+2*X$dA1_3[i+1]+X$dA1_4[i+1])/6+X$Bolus[i+1]/(60/timestep)
X$A2[i+1] <- X$A2[i]+(X$dA2_1[i+1]+2*X$dA2_2[i+1]+2*X$dA2_3[i+1]+X$dA2_4[i+1])/6
X$A3[i+1] <- X$A3[i]+(X$dA3_1[i+1]+2*X$dA3_2[i+1]+2*X$dA3_3[i+1]+X$dA3_4[i+1])/6
X$Ae[i+1] <- X$Ae[i]+(X$dAe_1[i+1]+2*X$dAe_2[i+1]+2*X$dAe_3[i+1]+X$dAe_4[i+1])/6
X$Cp[i+1] <- X$A1[i+1]/V1/1000
X$Ce[i+1] <- X$Ae[i+1]/V1/1000
X$C1[i+1] <- X$A1[i+1]/V1/1000
X$C2[i+1] <- X$A2[i+1]/V2/1000
X$C3[i+1] <- X$A3[i+1]/V3/1000
}
X <- X[-1,]
yl <- c(0, ifelse(input$ylim==0, max(X$Ce), ifelse(input$ylim<max(X$Ce), input$ylim, max(X$Ce))))
par(mar=c(5, 5, 2, 1), las=1, cex.lab=1.5)
plot(tx, X$Ce, type="n", ylim=yl, xlab="Time", ylab="Effect concentratoin [ng/ml]")
lines(tx, X$Ce, lwd=7)
abline(h=1.0, lty=3)
abline(h=2.0, lty=3, col="red", lwd=2)
abline(v=seq.POSIXt(tx[1], tail(tx, 1)+100, by=15*60), lty=3)
if(input$Cont_conc > 0){
x_ivPCA <- as.numeric(input$ivPCAstart)
abline(v=x_ivPCA, lty=3, col="yellow")
text(x_ivPCA, par()$usr[4], "ivPCA", pos=3, xpd=TRUE, cex=1.6)
}
})
output$info <- renderText({
xy_str <- function(e) {
if(is.null(e)) return("Click figure\n")
click_time <- format(as.POSIXlt(as.numeric(e$x), origin="1970-01-01"), "%H:%M")
sprintf("Ce is %.4f at %s", e$y, click_time)
}
paste0(
xy_str(input$plot_click)
)
})
}
shinyApp(ui = ui, server = server)