こんなことをした。
mikuhatsune.hatenadiary.com
集中治療学会が、人工呼吸器を要している患者、ECMOをしている患者、ECMOで死亡した患者、ECMOから回復した患者、など日ベースで公開してる。
これに、毎日の感染者や死亡者のデータをくっつけて、SIRを使って人工呼吸器を要する患者やECMOを要する患者を推定する。
Japan Coronavirus: 9,231 Cases and 190 Deaths - Worldometer
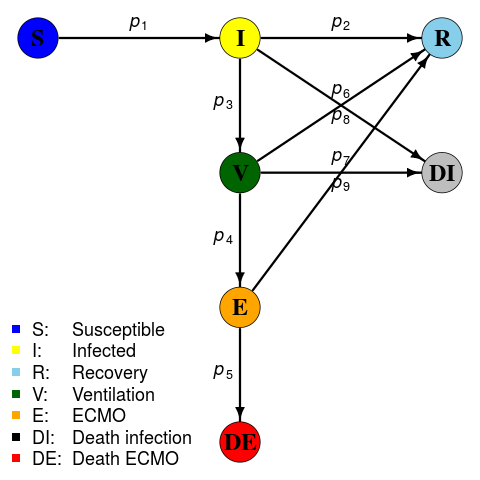
SIRモデルとしてはこんなのを想定する。
SIRは普通にSIRモデルだが、Iから人工呼吸器管理になる人V、VからECMO管理になる人E、ECMOから死亡する人DEとして
Iから死亡する人はDIとすると、推移にパラメータ は9つ必要で
は9つ必要で







となる。ただし とする。
とする。
最初の人口は1.2億人で始める。
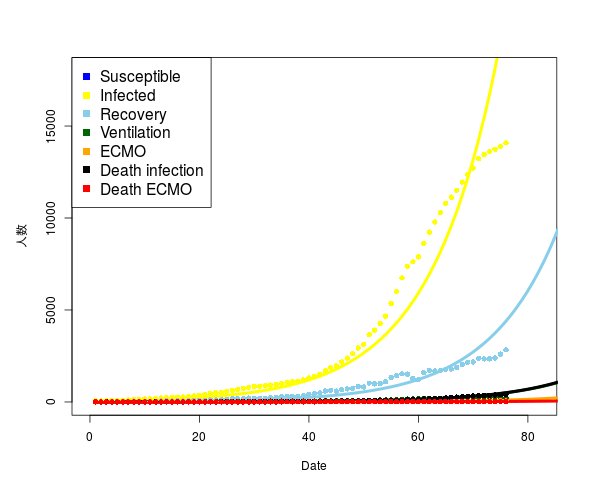
5月5日までのデータで既にかなりずれているようにも思えるが、SIRモデルの簡潔さからいうと仕方ないように思う。
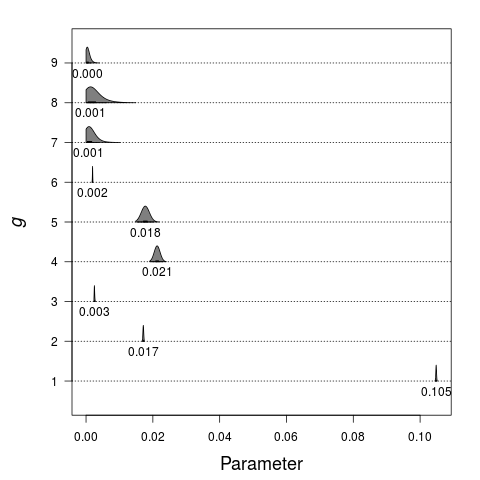
 はこんな感じになった。例えば
はこんな感じになった。例えば は人工呼吸器からECMOにいくパラメータで、
は人工呼吸器からECMOにいくパラメータで、 はECMOから死亡にいくパラメータだが、これは1日あたりなので、適当に積分したらいいのだろうか。
はECMOから死亡にいくパラメータだが、これは1日あたりなので、適当に積分したらいいのだろうか。
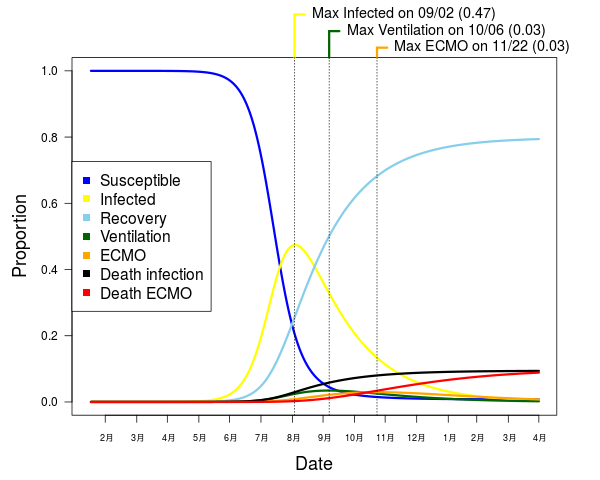
365日後まで推定すると、感染者(軽症も含む)のピークは9月2日にきて、人工呼吸器を要する患者のピークは10月6日、ECMOを行なっている患者のピークは11月22日にくる。集中治療としては10-11月中が最大の山場、となるのかもしれない。
mat <- read.csv("japan.csv", stringsAsFactors=FALSE)
ecmo <- merge(l222[,c("date", "全国")], merge(l555[,c("date", "全国")], l333, by.x="date", by.y="date"), by.x="date", by.y="date")
colnames(ecmo) <- c("date", "ECMOall", "ventilation", "ECMOrecovery", "ECMOdeath", "ECMOdoing")
mat$Date <- gsub("Apr ", "04-", gsub("Mar ", "03-", sprintf("2020-%s", gsub("Feb ", "02-", mat$Date))))
m <- as.data.frame(merge(ecmo, mat, by.x="date", by.y="Date"))
Pop <- 120000000
DE <- m$ECMOdeath
DI <- m$Total.Deaths - DE
E <- m$ECMOdoing
I <- m$Total.Cases
V <- m$ventilation - E
R <- I - m$Active.Cases
S <- Pop - (I + R + E + V + DI + DE)
N <- cbind.data.frame(S=S, I=I, R=R, V=V, E=E, DI=DI, DE=DE)
library(igraph)
G <- graph_from_edgelist(rbind(c(1,2),c(2,3),c(2,4),c(2,6),c(4,5),c(4,3),c(4,6),c(5,7),c(5,3)))
G <- graph_from_edgelist(rbind(c(1,2),c(2,3),c(2,4),c(4,5),c(5,7),c(2,6),c(4,6),c(4,3),c(5,3)))
lmat <- layout.norm(rbind(c(-1,0),c(0,0),c(1,0),c(0,-1),c(0,-2),c(1,-1),c(0,-3)))
pmat <- mapply(function(z) colMeans(lmat[get.edgelist(G)[z,],]), seq(nrow(get.edgelist(G))))
ppos <- c(3, 3, 2, 2, 2, 3, 3, 1, 1)
V(G)$label <- c("S", "I", "R", "V", "E", "DI", "DE")
V(G)$label.cex <- 2
V(G)$label.font <- 2
V(G)$label.color <- "black"
V(G)$size <- 20
V(G)$color <- cols
V(G)$color[6] <- "grey"
E(G)$color <- "black"
E(G)$width <- 3
par(mar=c(0, 0, 0, 0))
plot(G, layout=lmat)
legend("bottomleft", legend=sprintf("%s:\t%s", c("S", "I", "R", "V", "E", "DI", "DE"), names(cols)), pch=15, col=cols, bty="n", cex=1.5)
for(i in 1:9){
txt <- as.expression(substitute(italic(p[x]), list(x=i)))
text(pmat[1, i], pmat[2, i], txt, xpd=TRUE, pos=ppos[i], cex=1.5)
}
library(rstan)
rstan_options(auto_write=TRUE)
options(mc.cores=parallel::detectCores())
code <- "
data{
int<lower=1> T;
int<lower=1> Tf;
int<lower=1> N; // population
int<lower=0> S0[7]; //S, I, R, V, E, DI, DE
int<lower=0> S[T, 7]; //S, I, R, V, E, DI, DE
}
parameters{
vector<lower=0, upper=0.5>[9] p;
}
transformed parameters{
vector<lower=0>[7] y[T+Tf];
y[1] = to_vector(S0);
for(i in 1:(T+Tf-1)){
y[i+1][1] = y[i][1] - p[1]*y[i][1]*y[i][2]/sum(y[i]);
y[i+1][2] = y[i][2] + p[1]*y[i][1]*y[i][2]/sum(y[i]) - (p[2]+p[3]+p[6])*y[i][2];
y[i+1][3] = y[i][3] + p[2]*y[i][2]+p[8]*y[i][4]+p[9]*y[i][5];
y[i+1][4] = y[i][4] + p[3]*y[i][2] - (p[4]+p[7]+p[8])*y[i][4];
y[i+1][5] = y[i][5] + p[4]*y[i][4] - (p[5]+p[9])*y[i][5];
y[i+1][6] = y[i][6] + p[6]*y[i][2] + p[7]*y[i][4];
y[i+1][7] = y[i][7] + p[5]*y[i][5];
}
}
model{
for(i in 1:T){
S[i,] ~ multinomial(y[i]/sum(y[i]));
}
}
"
m0 <- stan_model(model_code=code)
standata <- list(T=nrow(N), S=N, S0=unlist(N[1,]), N=Pop, Tf=365)
fit <- sampling(m0, standata, iter=7000, warmup=5000, chain=4)
ex <- extract(fit, pars=head(fit@model_pars, -1))
alpha <- 0.05
cia <- c(alpha/2, 0.5, 1-alpha/2)
m <- abind::abind(mapply(function(z) apply(ex$y[,,z], 2, quantile, cia), seq(dim(ex$y)[3]), SIMPLIFY=FALSE), along=3)
p <- apply(ex$p, 2, quantile, cia)
cols <- c("Susceptible"="blue", "Infected"="yellow", "Recovery"="skyblue", Ventilation="darkgreen", ECMO="orange", "Death infection"="black", "Death ECMO"="red")
library(vioplot)
par(mar=c(4.5, 5, 2, 2), cex.lab=1.5)
vioplot(as.data.frame(ex$p), ylab=as.expression(substitute(italic(g))), xlab="Parameter", horizontal=TRUE, side="right", colMed=NA, las=1)
text(p[2,], seq(ncol(p)), sprintf("%.3f", p[2,]), pos=1)
abline(h=seq(ncol(p)), lty=3)
xl <- c(0, 82)
yl <- c(0, 18000)
par(mar=c(4.5, 5, 4, 3))
matplot(m[2,,-1], type="l", col=cols[-1], xlim=xl, ylim=yl, lwd=4, lty=1, xlab="Date", ylab="人数")
for(i in 2:ncol(N)){
points(N[,i], pch=16, col=cols[i])
}
legend("topleft", legend=names(cols), col=cols, pch=15, xpd=TRUE, ncol=1, cex=1.3)
xl <- c(0, dim(m)[2])
yl <- c(0, max(m[2,,-1]))
matplot(m[2,,-1], type="l", col=cols[-1], xlim=xl, ylim=yl, lwd=3, lty=1, xlab="", ylab="人数")
for(i in 2:ncol(N)){
points(N[,i], pch=16, col=cols[i])
}
Mday <- apply(m[2,,], 2, which.max)
xl <- c(0, dim(m)[2])
yl <- c(0, 1)
xd <- 10
yd <- 0.03 + 0.05*(0:2)
d <- seq(as.Date("2020-02-15"), by="day", length.out=dim(m)[2])
r <- rle(format(d, "%h"))
par(mar=c(4.5, 5, 4, 3))
matplot(m[2,,]/Pop, type="l", col=cols, xlim=xl, ylim=yl, lwd=3, lty=1, xlab="Date", ylab="Proportion", xaxt="n", las=1, cex.lab=1.5)
axis(1, at=cumsum(r$lengths), labels=r$values, cex.axis=0.75)
legend("left", legend=names(cols), col=cols, pch=15, xpd=TRUE, ncol=1, cex=1.3)
j <- 0
pa <- par()$usr
for(i in c(5, 4, 2)){
j <- j + 1
txt <- sprintf("Max %s on %s (%.2f)", names(cols)[i], format(d[Mday[i]], "%m/%d"), m[2, Mday[i], i]/Pop)
segments(Mday[i], pa[4], y1=pa[4]+yd[j], xpd=TRUE, col=cols[i], lwd=3)
segments(Mday[i], pa[4]+yd[j], x1=Mday[i]+xd, xpd=TRUE, col=cols[i], lwd=3)
text(Mday[i]+xd, pa[4]+yd[j], txt, xpd=TRUE, pos=4, cex=1.2)
abline(v=Mday[i], lty=3)
}
library(stringr)
country <- "japan"
html <- sprintf("https://www.worldometers.info/coronavirus/country/%s", country)
h <- paste(readLines(html), collapse="")
s0 <- str_extract_all(h, '<script type="text/javascript">.*?</script>')[[1]]
s1 <- mapply(function(z) str_extract_all(z, " categories: \\[.*?\\]"), s0)
s2 <- lapply(mapply(function(z) str_replace(str_replace_all(z, "categories|[\":\\[\\]]", ""), "^ +", ""), s1), unique)
s3 <- unname(sapply(s2, strsplit, ","))
d1 <- mapply(function(z) str_extract_all(z, "data: \\[.*?\\]"), s0)
d2 <- lapply(mapply(function(z) str_replace(str_replace_all(z, "data|[\":\\[\\]]", ""), "^ +", ""), d1), unique)
d3 <- unname(lapply(lapply(d2, strsplit, ","), lapply, as.numeric))
n1 <- mapply(str_extract_all, s0, " title: \\{\\s+text: '.+?'")
n2 <- unname(rapply(n1, function(z) str_remove_all(str_extract(z, "text: '.+?'"), "text: |'"), how="replace"))
mat <- cbind.data.frame(s3[[1]], d3[[2]][[1]], d3[[3]][[1]], d3[[4]][[1]], d3[[5]][[1]], d3[[6]][[1]], d3[[7]][[1]], d3[[8]][[1]])
colnames(mat) <- c("Date", n2[[2]][[1]], n2[[3]][1], n2[[4]][1], n2[[5]][1], n2[[6]][1], n2[[7]][1], "Recoveries")
write.table(mat, sprintf("%s.csv", country), sep=",", row.names=FALSE)











