結論から言うと、substitute 関数を使って数式を記述し、atop を分子と分母について2行にわたって記述したいテキストに対してそれぞれ使う。ただし、分数表記でいうと微妙に高さがずれるので、atop は中央の括線を書かないので気にしない、ということにすればなんとかなる。
こんな感じの図を書きたいと思った。
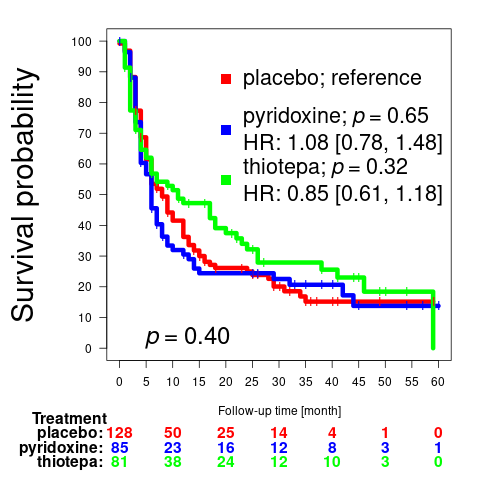
カプランマイヤーの生存曲線だが、3群比較で、Cox 比例ハザード比を用いて、対照となる群に対して残りの2群のp値やハザード比、95%信頼区間をlegend に詳細に書きたいのだが、pはイタリックでpにしたいし、1行に全部書いていたら長くなりすぎるため、2行にしたかった。
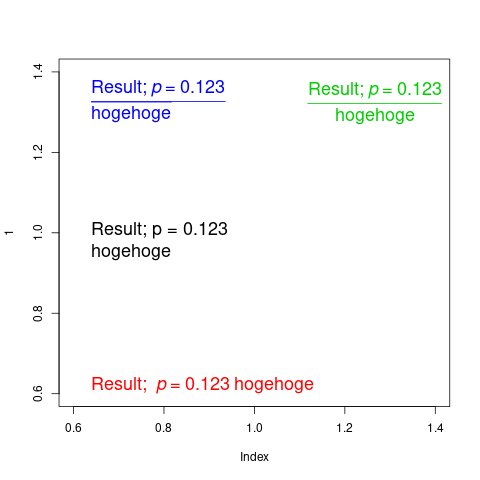
ここで、最も簡単に左揃えで2行にテキストを収めるには、Result; p = 0.123\nhogehoge と改行コードを含めてテキストオブジェクトを作ることだが、これをするとp が数式扱いされず、イタリックにならない。これでは見栄えが許されない。
簡単に数式を含むテキストを作成するには、substitute 関数を使えばよい。しかし、substitute に改行コードを含めても、残念ながら改行してくれない。
ということで、2行にする場合には、無理やり分数として記述すればよい。substitute で分数を記述する場合には、中央の括線を書く場合にはover、書かない場合にはatop で書ける。しかし、何も考えずにatop を使った場合には、分子と分母にくるテキストの長さが異なる場合、それぞれ中央寄せになってしまう。
なので、分子だけをatop で記述し、次に分母だけをatop で再度記述することで、分子と空の分母、空の分子と分母のテキストをそれぞれ書くことになり、それぞれが勝手に左寄せになる、ということで無理やり左寄せ2行テキストを書いた。
しかしこれでは、テキストの上下配置が微妙に異なることがあり、検証用にover で書いてみたところ、括線がずれていたので気持ち悪さが残るが、atop なら透明なので気にしないことにすれば、目的は達成される。
ちなみに空の数式テキストはphantom(0) である。
latex2exp もあるよ、とは教えてもらったが、latex2exp はmatrix などの記法に対応していないので使えなかった。
library(survival) library(prodlim) dat <- cbind.data.frame(treatment=bladder1$treatment, time=bladder1$stop - bladder1$start, event=ifelse(bladder1$status > 0, 1, 0)) f <- coxph(Surv(time, event) ~ treatment, dat) g <- summary(f) s <- survfit(Surv(time, event, type="right") ~ treatment, data=dat) s <- prodlim(Hist(time, event) ~ treatment, data=dat) cols <- c("red", "blue", "green") names(cols) <- levels(dat$treatment) xl <- c(0, 60) yl <- c(0, 1) at.t <- seq(0, max(xl), by=10) axis1.at <- seq(0, max(xl), by=5) axis2.at <- seq(0, max(yl), by=0.1) par(mar=c(3, 4, 2, 2), las=1) plot(s, atrisk.labels=sprintf("%s: ", names(cols)), xlim=xl, ylim=yl, xlab="Follow-up time [month]", ylab="", col=cols, atrisk.title="Treatment", axis1.at=axis1.at, axis1.labels=axis1.at, axis2.at=axis2.at, axis2.labels=axis2.at*100, atrisk.at=at.t, atrisk.font=2, atrisk.cex=1.3, legend.x="bottomright", legend.cex=1, legend=FALSE, confint=FALSE, marktime=TRUE, background=TRUE, background.horizontal=NA, logrank=TRUE, lwd=6) legend("bottomleft", legend=substitute(italic(p)~"="~x, list(x=sprintf("%.2f", g$logtest["pvalue"]))), bty="n", cex=2) mtext("Survival probability", 2, line=1.5, srt=90, las=3, cex=2.5) txt <- list(as.expression(c( sprintf("%s; reference", names(cols)[1]), substitute(atop(x1~italic(p)~"="~x2, phantom(0)), list(x1=sprintf("%s;", names(cols)[2]), x2=sprintf("%.2f", g$coefficients[1,5]))), substitute(atop(phantom(0), x3), list(x3=sprintf("HR: %.2f [%.2f, %.2f]", g$conf.int[2,1], g$conf.int[2,3], g$conf.int[2,4]))) )), as.expression(c( "", substitute(atop(phantom(0), x3), list(x3=sprintf("HR: %.2f [%.2f, %.2f]", g$conf.int[1,1], g$conf.int[1,3], g$conf.int[1,4]))), substitute(atop(x1~italic(p)~"="~x2, phantom(0)), list(x1=sprintf("%s;", names(cols)[3]), x2=sprintf("%.2f", g$coefficients[2,5]))) )) ) for(j in seq(txt)){ legend("topright", legend=txt[[j]], col=cols, bty="n", cex=1.8, ncol=1, pch=15) } pv <- 0.123 txts <- list( as.expression(sprintf("Result; p = %.3f\nhogehoge", pv)), as.expression(substitute("Result; "~italic(p)~"="~x1~"\nhogehoge", list(x1=sprintf("%.3f", pv)))), as.expression(substitute(over(x1~italic(p)~"="~x2, x3), list(x1="Result;", x2=sprintf("%.3f", pv), x3="\nhogehoge"))), list( as.expression(substitute(over(x1~italic(p)~"="~x2, phantom(0)), list(x1="Result;", x2=sprintf("%.3f", pv)))), as.expression(substitute(over(phantom(0), x3), list(x3="hogehoge"))) ) ) pos <- c("left", "bottomleft", "topright", "topleft") cols <- seq(pos) plot(1, type="n") for(i in seq(txts)){ for(j in seq(txts[[i]])){ legend(pos[i], legend=txts[[i]][[j]], bty="n", cex=1.5, text.col=cols[i]) } }