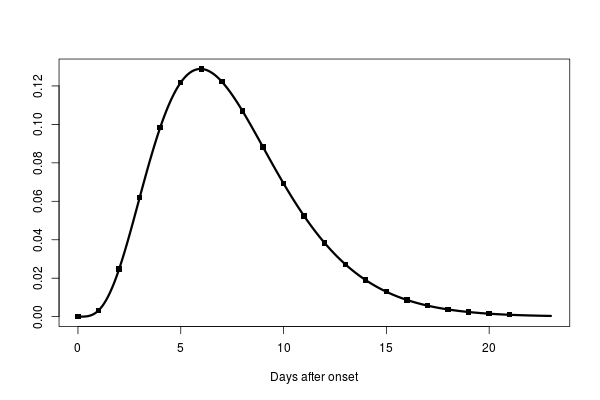
最初に武漢で肺炎が発生したときに、88症例について感染履歴を聴取して、ワイブル分布で潜伏期間を推定すると平均6.4日(95% credible interval (CI): 5.6–7.7)、潜伏期間の幅は2.1から11.1日(2.5th to 97.5th percentile)だった、という。
論文ではワイブル分布のほかに、ガンマ分布、対数正規分布で推定して、looicでもっともよかったのがワイブル分布だった、と言っている。
supplemental にスクリプトがあったのでぱくってやってみる。
結果としてはだいぶずれたような気がする。
| weibull | gamma | lognormal | |
| 2.5% | 4.629226 | 5.531133 | 5.174367 |
| 50% | 6.828260 | 6.353485 | 6.070993 |
| 97.5% | 9.616947 | 7.583359 | 7.347166 |
| looIC | 480.119496 | 524.649064 | 586.254024 |
前処理にtidyverseがうざすぎるのでこちらで前処理したデータを置いておく。
library(rstan) library(loo) rstan_options(auto_write=TRUE) options(mc.cores=parallel::detectCores()) # tidyverse がうざいので加工済みデータを置いておく input <- list(N=88, tStartExposure=c(-2,-18,-18,10,3,8,12,13,-18,-18,8,10,-11,-18,-18,-18,12,-18,-18,-18,-18,-18,-18,11,11,-18,-18,-18,-18,-18,12,12,15,15,-18,-18,-18,-18,19,-18,-18,-18,-18,-18,-18,-18,18,-18,-18,6,-18,-18,-18,9,-18,11,-18,-18,-18,-18,-18,-18,-18,-18,-18,-18,19,-18,-18,-18,-18,-18,-18,-18,13,13,-18,-18,-18,-18,-18,-18,13,-18,-18,-18,-18,-18), tEndExposure=c(3,12,3,11,4,16,16,16,15,15,16,11,9,2,12,17,15,17,18,13,16,11,18,14,18,20,12,10,17,15,15,14,17,20,18,13,23,23,22,20,21,21,21,15,21,17,20,18,20,7,20,20,20,20,18,22,22,18,18,18,9,20,18,22,23,19,19,22,22,13,22,22,23,23,17,17,22,18,18,22,18,20,15,20,19,23,22,22), tSymptomOnset=c(3,15,4,14,9,16,16,16,16,16,16,14,10,10,14,20,19,19,20,20,17,13,19,19,20,21,18,18,18,16,20,16,20,20,19,17,24,24,23,21,23,23,23,21,22,24,24,19,21,14,23,23,21,20,21,22,23,19,23,21,13,22,24,25,25,25,25,24,24,21,23,23,24,25,18,18,23,22,22,24,24,25,22,25,25,24,25,25) ) # 3つの確率分布を一気に作る dists <- c("weibull", "gamma", "lognormal") code <- sprintf(" data{ int<lower=1> N; vector[N] tStartExposure; vector[N] tEndExposure; vector[N] tSymptomOnset; } parameters{ real<lower=0> par[2]; vector<lower=0, upper=1>[N] uE; // Uniform value for sampling between start and end exposure } transformed parameters{ vector[N] tE; // infection moment tE = tStartExposure + uE .* (tEndExposure - tStartExposure); } model{ // Contribution to likelihood of incubation period target += %s_lpdf(tSymptomOnset - tE | par[1], par[2]); } generated quantities { // likelihood for calculation of looIC vector[N] log_lik; for (i in 1:N) { log_lik[i] = %s_lpdf(tSymptomOnset[i] - tE[i] | par[1], par[2]); } } ", dists, dists) names(code) <- dists m0 <- mapply(stan_model, model_code=code) fit <- mapply(sampling, m0, list(input), iter=10000, warmup=3000, chain=4) ps <- mapply(function(z) extract(z)$par, fit, SIMPLIFY=FALSE) # 確率分布のパラメータから得られる解析的な平均値 means <- cbind(ps$weibull[,1]*factorial(1+1/ps$weibull[,2]+1), ps$gamma[,1] / ps$gamma[,2], exp(ps$lognormal[,1])) alpha <- c(0.025, 0.5, 0.975) res <- apply(means, 2, quantile, alpha) ll <- mapply(function(z) loo(extract_log_lik(z))$looic, fit) rbind(res, looIC=ll)