この記事は
R Advent Calendar 2016
Stan Advent Calendar 2016
ごちうさ Advent Calendar 2016
まんがタイムきらら Advent Calendar 2016
の25日目の担当記事です。
C91 で声優統計ネタとして声優力を推定します。声優力とはなんぞや、という話ですが、ある声優がアニメに出演するとき、主役だったりメインヒロインだったりすると、上位にキャストされると思います。そのキャストされるのがどれだけ上位か、というのをデータから得て、声優力の推定をします。
例えば総勢 人の声優が出演するとき、その
人の声優が出演するとき、その 番目に名前があったとしたら、
番目に名前があったとしたら、 として声優力を[0,1] のデータにします。1 に近いと上位にいて、0に近いと末尾に名前があることに相当します。
として声優力を[0,1] のデータにします。1 に近いと上位にいて、0に近いと末尾に名前があることに相当します。
これは[0,1] にしたかったので適当な変換になります。[0,1] にすると、 がベータ分布からサンプリングされるだろうと勝手に仮定できます。ベータ分布を決めているパラメータは、shape parameter 1 と2 の
がベータ分布からサンプリングされるだろうと勝手に仮定できます。ベータ分布を決めているパラメータは、shape parameter 1 と2 の ,
,  であり、この分布の平均値は
であり、この分布の平均値は となります。サンプリングは
となります。サンプリングは

となります。
これを2008年から2016年まで、1年を3ヶ月ずつ冬春夏秋クールの時系列に分けて推定する。
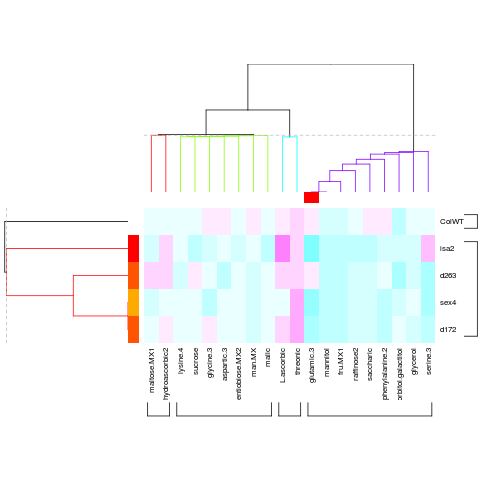
ごちうさ声優陣である佐倉綾音、水瀬いのり、種田梨沙、佐藤聡美、内田真礼、茅野愛衣、村川梨衣、徳井青空、早見沙織の声優力をみてみましょう。
村川梨衣以外は、全期間で割と高い声優力を有しているようなクラスターに属した。村川梨衣は2013年以降に声優力が高いクラスター、つまり結構若手なクラスターに属した。

村川梨衣以外のごちゃっとしたクラスターを拡大して検討してみる。
左にあるクラスタリングを適当に6個に分けた。
下から、ピンクと茶色のクラスターは、2008年から2016年までぎっちりと高い声優力を保って出演を続けている声優たちである。ピンクのほうは2016年直近もよく出ているが、茶色は2016年に近い年代で白(出演なし)が目立つところで差が出ている。
ピンクのクラスターには戸松遥、豊崎愛生、井上麻里奈、悠木碧、釘宮理恵、伊藤静、沢城みゆき、福園美里、花澤香菜らが属した。誰が聞いてもわかるし納得の面々だと思う。個人的には井上麻里奈、伊藤静がいるのが超うれしい。
茶色には早見沙織が属した。はやみんは2016年もよく出ているが、どちらかというと2008年ころの出演がないの茶色に属したようである。
濃い緑のクラスターは、2011年から2012年ころから出演しはじめた声優が属している。水瀬いのり、種田梨沙らが属した。黄色の点線は種田梨沙のデビューということで引いている。
声優統計本誌では、種田梨沙に近くて種田梨沙の声優力を模倣してくれるであろう声優をピックアップし、2017年に推して行こうという話が書かれている。
青緑のクラスターは、2008年から出演しているが、直近ではかなり白が目立つクラスターとなった。ただし、2016年直近は出演情報がデータベースにきちんと登録されていない可能性があるため、「本当にチョイ役だけど載っていない」とかだと空白になってしまう。
青のクラスターは、2010年から2011年ころから出演しており、直近では黒色が濃い(声優力が高い)クラスターとなった。ここでは内田真礼、徳井青空、佐倉綾音、茅野愛衣など、若手超実力派といっても過言ではない声優が属した。茅野愛衣がここにくるのは予想外だったが、思い返してみると茅野愛衣が自分の中で爆発したのは2011年のあの花のめんま役だったので、意外とまだ若手〜中堅といってもいいのだろう。ただしオレの中での存在感はやばい。
紫のクラスターは、2008年から継続的に出演しているが、2012年以前は色が濃く、2012年以降は色が薄いもしくは白抜けが目立つ感じのクラスターとなった。佐藤聡美はここに属した。思い返せば2009年のけいおん!からいるので、かなり古参である。

実際に時系列でプロットしてみるとほとんどが0.5 周辺でうろうろしているだけのような気がして、本当に声優力が[0,1]でいい感じに推定されているのかはちょっとよくわからない。ベータ分布は無情報事前分布だと平均が0.5 から始まるので0.5 周辺から抜け出さないのか、そもそもモデルがアレなのか…
グラフ内の線の色はキャラのイメージカラー、凡例内の声優の色はクラスター色と対応している。

一応収束したモデル
data{
int<lower=0> n_time;
int<lower=0> n_kr[n_time];
int<lower=0> n_cast;
real<lower=0, upper=1> p[n_cast];
}
parameters{
real<lower=0> alpha[n_time];
real<lower=0> beta[n_time];
real<lower=0> s_a;
real<lower=0> s_b;
}
model{
for(i in 3:n_time){
alpha[i] ~ lognormal(2*alpha[i-1]-alpha[i-2], s_a);
beta[i] ~ lognormal(2*beta[i-1]-beta[i-2], s_b);
}
for(i in 1:n_time){
if (n_kr[i] > 0) {
for(j in 1:n_kr[i]){
p[sum(n_kr[1:(i-1)])+j] ~ beta(alpha[i], beta[i]);
}
}
}
}
import os
import re
import progressbar
wd = "/shoboi/"
files = os.listdir(wd)
cast = '<div class="title">キャスト</div>'
cvre = re.compile('="nofollow">.{1,}?</a>')
widgets = ["progress:", Percentage(), Bar()]
pbar = ProgressBar(maxval=len(files), widgets=widgets).start()
table = '</table>'
w0 = open("cast.txt", "w")
w0.write("\t".join(["title", "start", "cv"]) + "\n")
res = []
for f in range(len(files)):
g = open(wd + files[f], "rU")
res0 = []
res1 = "NA"
cvlist =[]
flag = 0
for tmp in g:
if cast in tmp:
flag = 1
if table in tmp:
flag = 2
if 'しょぼいカレンダー</title>' in tmp:
res0 += [ re.compile(".+</title>").findall(tmp)[0][9:-38] ]
if '<tr><th>放送期間</th><td>' in tmp:
day = re.compile("\d{4}-\d{1,}").findall(tmp)
if len(day) > 0:
res1 = day[0]
else:
res1 = "NA"
if flag == 1:
r = cvre.findall(tmp)
if len(r) > 0:
cvlist += [ r[0][12:-4].rstrip() ]
if flag == 2:
pass
res = [ " ".join(res0), res1, ",".join(cvlist) ]
w0.write("\t".join(res) + "\n")
print f
w0.close()
dat <- read.delim("cast.txt", stringsAsFactors=FALSE)
cv <- mapply(strsplit, dat$cv, ",")
names(cv) <- NULL
cvs <- unique(unlist(cv))
as.Date(paste(dat$start, "01", sep="-"))
d <- as.Date(paste0(dat$start, "-01"))
kr <- seq.Date(min(d, na.rm=TRUE), max(d, na.rm=TRUE), by="quarter")
res <- vector("list", length(cvs))
names(res) <- cvs
pb <- txtProgressBar(min=1, max=length(cvs), style=3)
for(i in seq(cvs)){
setTxtProgressBar(pb, i)
j <- which(mapply(function(z) cvs[i] %in% z, cv))
rank0 <- mapply(function(z) match(cvs[i], z), cv[j])
all0 <- sapply(cv[j], length)
top0 <- all0 - rank0 + 1
mat <- cbind(rank=rank0, all=all0, top=top0)
y <- split(as.data.frame(mat), cut(d, kr)[j])
res[[i]] <- y
}
hoge <- mapply(function(z) sum(sapply(z, nrow)), res)
res0 <- res[ hoge > 4 ]
fidx <- read.csv("sex.csv", stringsAsFactors=FALSE)$sex
res0 <- res0[fidx == "女性" & !is.na(fidx)]
n_kr <- mapply(function(z) sapply(z, nrow), res0)
data.frame(name=names(res0), sex=prof$sex[match(names(res0), prof$name)])
bar <- do.call(rbind, res0[[i]])
y <- sapply(res0[[i]], nrow)
kr_idx <- rep(match(names(y)[y>0], as.character(kr)), y[y>0])
n_kr <- sapply(res0[[i]], nrow)
kr0 <- head(tail(kr, length(kr)-rle(n_kr>0)$length[1]), -1)
n_kr0 <- n_kr[as.character(kr0)]
rstan_options(auto_write = TRUE)
options(mc.cores = parallel::detectCores())
model <- stan_model("model01.stan")
stan_data <- list(n_time=length(n_kr0), n_kr=n_kr0, n_cast=nrow(bar), N=bar$top)
fit <- sampling(model, data=stan_data, chains=3, warmup=30, iter=30, seed=1234)
bm0 <- ex$alpha/(ex$alpha + ex$beta)
bm <- apply(bm0, 2, median)
mv <- tapply(stan_data$p, rep(seq(stan_data$n_kr), stan_data$n_kr), median)
mv <- replace(rep(0, length(n_kr0)), as.numeric(names(mv)), mv)
alpha <- 0.05
cols <- c("black", "blue")
ci <- apply(bm0, 2, quantile, c(alpha/2, 1-alpha/2))
xy <- cbind(bm, mv)
par(mar=c(4, 4.5, 2, 2), cex.lab=1.5, cex.axis=1.5)
matplot(xy, type="n", ylim=c(0, 1), xlab="", ylab="Parameter", las=1, lty=c(1,3), xaxt="n")
polygon(c(1:ncol(ci), ncol(ci):1), c(ci[1,], rev(ci[2,])), col=grey(0.9), border=NA)
for(k in 1:ncol(xy)) lines(1:nrow(xy), xy[,k], type="o", pch=15, lwd=3, col=cols[k], lty=c(1,3)[k])
ymd <- do.call(rbind, strsplit(names(n_kr0), "-"))
colnames(ymd) <- c("y", "m", "d")
ymd <- as.data.frame(ymd, stringsAsFactors=TRUE)
season <- c("冬", "春", "夏", "秋")
xd <- 0.0
yd <- 0.03
for(i in seq(levels(ymd$y))){
tmp <- which(c(ymd$y) == i)
x0 <- tmp[1]
x1 <- tail(tmp, 1)
xs <- seq(length(n_kr0))
segments(xs[x0]+xd, par()$usr[3]-yd, xs[x1]-xd, lwd=2, xpd=TRUE)
text(mean(xs[tmp]), par()$usr[3]-yd, levels(ymd$y)[i], xpd=TRUE, pos=1, cex=1.2)
text(xs[tmp], par()$usr[3]-yd*3.2, season[c(ymd$m)[tmp]], xpd=TRUE, pos=1, cex=1.2)
}
abline(v=cumsum(head(rle(c(ymd$y))$lengths, -1))+0.5, lty=3)
legend("bottomright", legend=c("Estimate", "Observation"), lty=c(1,3), lwd=3, col=cols, bg="white", cex=1.5)
gochiusa <- c("佐倉綾音", "水瀬いのり", "種田梨沙", "佐藤聡美", "内田真礼", "茅野愛衣", "村川梨衣", "徳井青空", "早見沙織")
gochiusa_col <- c("plum1", "lightskyblue", "blueviolet", "aquamarine3", "gold2", "orange", "red1", "blue1", grey(0.3))
h1 <- cut(h$rowDendrogram, h=6000)$lower[[1]]
h1 <- color_branches(h1, k=6)
h1 <- set(h1, "branches_lwd", 5)
h1attr <- rapply(h1, attributes)
clust_col <- h1attr[names(h1attr) == "edgePar.col"]
names(clust_col) <- h1attr[grep("label", names(h1attr))]
idx <- colnames(m0)[unlist(h1)]
layout(matrix(1:2, nc=2), widths=c(2, 10))
par(mar=c(0.1, 0.5, 0, 0.3))
plot(h1, horiz=TRUE, leaflab="none", yaxt="n", xaxs="i")
par(mar=c(5, 0.1, 4.5, 6))
image(seq(nrow(h$carpet)), seq(ncol(h$carpet[, idx])), h$carpet[,idx], col=cols, xaxt="n", yaxt="n", xlab="", ylab="")
cvy <- c(-0.6, -2, 2, 0.6, 0, 0)*10
tip.color <- replace(rep("black", length(idx)), match(gochiusa, idx), "blue")
text(par()$usr[2], seq(idx), idx, xpd=TRUE, pos=4, col=tip.color)
abline(v=which(rownames(h$carpet) == "2012-07-01")-0.5, lty=3, lwd=3, col="yellow")
ymd <- do.call(rbind, strsplit(rownames(m0), "-"))
colnames(ymd) <- c("y", "m", "d")
ymd <- as.data.frame(ymd, stringsAsFactors=TRUE)
season <- c("冬", "春", "夏", "秋")
xd <- 0.0
yd <- 2
for(i in seq(levels(ymd$y))){
tmp <- which(c(ymd$y) == i)
x0 <- tmp[1]
x1 <- tail(tmp, 1)
xs <- seq(nrow(h$carpet))
segments(xs[x0]+xd, par()$usr[3]-yd, xs[x1]-xd, lwd=2, xpd=TRUE)
text(mean(xs[tmp]), par()$usr[3]-yd, levels(ymd$y)[i], xpd=TRUE, pos=1)
text(xs[tmp], par()$usr[3]-yd*0, season[c(ymd$m)[tmp]], xpd=TRUE, pos=1)
}
G <- m0[, gochiusa]
G[G == 0] <- NA
par(mar=c(4, 4.5, 2, 2), cex.lab=1.5, cex.axis=1.5)
matplot(G, type="o", ylim=c(0, 1), xlab="", ylab="Parameter", col=gochiusa_col, las=1, lty=1, lwd=3, pch=15, xaxt="n")
ymd <- do.call(rbind, strsplit(names(n_kr0), "-"))
colnames(ymd) <- c("y", "m", "d")
ymd <- as.data.frame(ymd, stringsAsFactors=TRUE)
season <- c("冬", "春", "夏", "秋")
xd <- 0.0
yd <- 0.03
for(i in seq(levels(ymd$y))){
tmp <- which(c(ymd$y) == i)
x0 <- tmp[1]
x1 <- tail(tmp, 1)
xs <- seq(length(n_kr0))
segments(xs[x0]+xd, par()$usr[3]-yd, xs[x1]-xd, lwd=2, xpd=TRUE)
text(mean(xs[tmp]), par()$usr[3]-yd, levels(ymd$y)[i], xpd=TRUE, pos=1, cex=1.2)
text(xs[tmp], par()$usr[3]-yd*2.2, season[c(ymd$m)[tmp]], xpd=TRUE, pos=1, cex=1.2)
}
abline(v=cumsum(head(rle(c(ymd$y))$lengths, -1))+0.5, lty=3)
legend("bottomright", legend=gochiusa, col=gochiusa_col, text.col=clust_col[gochiusa], lwd=3, lty=1, pch=15, merge=TRUE, cex=1.2, bg="white")
がある。あるベース
に各キャラの効果
、グループ効果
があり、18 人の所属は
であるとする。18人のハイパーパラメータ
は
はディリクレ分布
程度が多く、収束しなかった。また、n_eff が全然なかった。
が何十万とかなって単純にμ's だと何倍人気になる、というのがわかりにくかったので、
の事後分布を各グループについて中央値を取って何倍人気に差があるか、にしている。すると2.5倍くらいμ's とAqours に人気の差があるようだった。